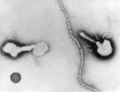
RNA病毒
编辑RNA病毒是一种以RNA(核糖核酸)为遗传物质的病毒。[1] 其核酸通常是单链RNA(ssRNA)。 但也可能是双链RNA。值得注意的由RNA病毒引起的的人类疾病包括埃博拉病毒病、非典型肺炎、狂犬病、普通感冒、流感、丙型肝炎、戊型肝炎、西尼罗河热、脊髓灰质炎和麻疹。
国际病毒分类委员会(ICTV)将RNA病毒分类为属于巴尔的摩分类病毒分类系统的第III组,第IV组或第V组的病毒。[2] 并不认为其在生命周期中含有DNA中间体的病毒为RNA病毒。以RNA为遗传物质的病毒,如果其复制周期中包含DNA中间体,则称为逆转录病毒,属于巴尔的摩分类的第VI组。值得注意的人类逆转录病毒包括HIV-1和HIV-2,这些是艾滋病的病因。
RNA病毒的另一个名称是核糖病毒,应该明确将逆转录病毒排除在外。[3]
目录编辑
- 3.1 正义链RNA病毒
- 3.2 双链RNA病毒
- 3.3 反义链RNA病毒
- 3.4 卫星病毒
- 4第III组——dsRNA病毒
- 5第四组——正义链ssRNA病毒
1 特征编辑
1.1 单链RNA病毒及其正反义链分类
RNA病毒可以根据其RNA的正反义链或极性进一步分为正义链、反义链和双义RNA病毒。正义链病毒的RNA与mRNA相似,因此可以被宿主细胞直接翻译。反义链病毒的RNA与mRNA互补,因此在翻译前必须通过依赖RNA的RNA聚合酶转录为正义链RNA。因此,纯化的正义链病毒RNA虽然可能比整个病毒颗粒的传染性低,但可以直接引起感染。因为反义链RNA需要转录为正义链RNA,纯化的反义链病毒RNA本身不具有感染性;每个病毒粒子可以转录成多个正义链RNA。双义RNA病毒除了也从反义链翻译基因,其他方面类似于反义链RNA病毒。[4]
1.2 双链RNA病毒
双链(ds)RNA病毒代表多种病毒,病毒在宿主范围(人类,动物,植物,真菌和细菌)、基因组区段编号(一至十二)和病毒粒子组成(三角测量编号,衣壳层,突起,炮塔样顶点)等方面均有较大差异。其成员包括轮状病毒(全球公认的幼儿胃肠炎的最常见病因);小RNA病毒(全球公认的人类和动物粪便样本中最常见的病毒,无论是否有腹泻迹象);蓝舌病病毒(感染经济牛羊的一种重要病原体)。近年来,在原子和亚纳米水平上确定一些关键的病毒蛋白,并且在多种dsRNA病毒粒子的衣壳结构方面取得了显著进展,突出了这些病毒的结构和复制过程中的显著相似之处。[5]
1.3 突变率
与DNA病毒相比,RNA病毒通常具有较高的突变率,[5] 因为病毒RNA聚合酶缺乏DNA聚合酶的校对能力。 这就是很难制造有效的疫苗来预防由RNA病毒引起疾病的原因之一。[6]因为逆转录病毒的逆转录过程中出现的错误在病毒复制前就被嵌入到了两条DNA链中,所以即使其基因中间体可以整合到宿主基因组中,逆转录病毒也有很高的突变率(并且一旦整合就要接受宿主的基因校对)。[7]RNA病毒的某些基因对病毒复制周期很重要,并且不能耐受突变。例如,丙型肝炎病毒基因组中编码核心蛋白的区域高度保守,[8] 因为它包含一个参与内部核糖体进入位点的RNA结构。[9]
2 复制编辑
动物RNA病毒由ICTV分类。根据其基因组类型和复制方式,分为三组不同的RNA病毒:
- 双链RNA病毒(第III组)包含一至十多种不同的RNA分子,每种分子编码一种或多种病毒蛋白。
- 正义链ssRNA病毒(第IV组)的基因组直接用作mRNA,宿主核糖体将其翻译为单个蛋白质,由宿主和病毒蛋白质修饰,形成复制所需的各种蛋白质。其中之一包括依赖RNA的RNA聚合酶(RNA复制酶),以形成双链复制形式复制病毒RNA。反过来,这种dsRNA指导新病毒RNA的形成。
- 反义链ssRNA病毒(第V组)必须通过RNA复制酶复制其基因组以形成正义链RNA。这意味着病毒必须携带RNA复制酶。然后正义链RNA分子充当病毒mRNA,并通过宿主核糖体翻译成蛋白质。
逆转录病毒(第VI组)具有单链RNA基因组,但通常不被认为是RNA病毒,因为它们使用DNA中间体进行复制。逆转录酶是来自病毒本身的一种病毒酶,在病毒解除包被后,它将病毒RNA转化为互补的DNA链,复制后产生双链的病毒DNA分子。在使用病毒整合酶将DNA整合到宿主基因组中之后,编码基因的表达可导致新病毒粒子的形成。
3 分类编辑
已经证明RNA病毒的分类是一个难题。部分原因是这些基因组的高突变率。分类主要基于基因组的类型(双链、负、反义链或正义链)、基因数量和病毒组成。目前已识别出5个目47个科的RNA病毒。还有部分未下设的种和属。
类病毒和RNA卫星病毒与RNA病毒相关但是有所不同。这些病毒目前没有被归类为RNA病毒。
对数千种RNA病毒的研究表明,至少存在五种主要分类群:光滑病毒及其亲属群;小RNA病毒超群;阿尔法病毒超群和黄症病毒超群;dsRNA病毒和-ve链病毒。[10] 慢病毒群可能是所有剩余RNA病毒的基础。下一个主要的工作是划分小RNA超群和剩余的病毒。dsRNA病毒似乎是从+ve RNA祖先和dsRNA病毒内的-ve RNA病毒进化而来的。与病毒链最密切相关的是呼肠孤病毒科。
3.1 正义链RNA病毒
这是最大的一组RNA病毒群[11] 有30个科。人们试图将这些科按更高的目分组。这些提议基于对RNA聚合酶的分析,目前仍在考虑之中。迄今为止,由于怀疑单个基因是否适合确定进化支的分类,所以提出的建议尚未被广泛接受。
正义链RNA病毒的分类基于依赖RNA的RNA聚合酶。已经认可的三个组群:[12]
- Bymoviruses、豇豆花叶病毒、nepoviruses、野田病毒、小RNA病毒、马铃薯Y病毒、南方菜豆花叶病毒和一部分黄症病毒(甜菜西黄病毒和马铃薯卷叶病毒)——类picorna群(Picornavirata)。
- Carmoviruses、dianthoviruses、黄病毒、pestiviruses、statoviruses、番茄丛矮病毒、单链RNA噬菌体、丙型肝炎病毒和一部分黄症病毒(大麦黄矮病毒)——类黄酮群(Flavivirata)。
- 阿尔法病毒、carlaviruses、真菌传杆状病毒、大麦病毒、potexviruses、rubiviruses、烟草脆裂病毒、tricornaviruses、芜菁黄花叶病毒、苹果褪绿叶斑病毒、甜菜黄病毒和戊型肝炎病毒——α样群(Rubivirata)。
已经提议基于位于病毒复制中涉及的蛋白质的N末端附近的新结构域对α样(辛德比斯样)超群进行划分。[13] 提议划分的两组是:“altovirus”组包括(阿尔法病毒、furoviruses、戊型肝炎病毒、hordeiviruses、烟草花叶病毒、烟草脆裂病毒、tricornaviruses,可能还有rubiviruses);和“typovirus”组(苹果褪绿叶斑病毒、carlaviruses、potexviruses和芜菁黄花叶病毒)。
类阿尔法超群可以进一步分为三个分支:类rubi病毒、类tobamo病毒和类tymo病毒。[14]
其他的工作已经确定了五组正义链RNA病毒,分别包含4个目、3个目、3个目、1个目和一个目。[15] 这14个 目包含31个病毒科(包括17个植物病毒科)和48个属(包括30个植物病毒属)。该分析表明,阿尔法病毒和黄症病毒可以分为两个科,分别是托加病毒科和黄病毒科,但也表明其他分类学任务,如瘟病毒、丙型肝炎病毒、rubiviruses、戊型肝炎病毒和动脉病毒的分类能是不正确的。冠状病毒和圆环病毒可能是不同目的不同科,而不是目前分类的同一科的不同属。黄症病毒可能是两个科而不是一个科,苹果褪绿叶斑病毒可能不是一种黄化丝状病毒,而是痘病毒科的一个新属。
- 演化
基于对RNA聚合酶和解旋酶的分析,小RNA病毒的进化的进化可能追溯到真核生物的分化。[16] 他们推定的祖先包括细菌II组逆转录因子,HtrA蛋白酶家族和DNA噬菌体。
分体病毒与整体病毒的祖先有关,可能是从整体病毒的祖先进化而来的。[17]
低毒性病毒和杆菌状核糖核酸病毒可能分别与马铃薯病毒和南方菜豆花叶病毒有着共同的祖先。[17]
3.2 双链RNA病毒
这一分析还表明,dsRNA病毒相互之间并不密切相关,而是属于另外4科——双核糖核酸病毒科,囊状噬菌体科,分体病毒科和呼肠孤病毒科——和与正义链RNA病毒在同一亚门中的一类正义链ssRNA病毒的另外一科(整体病毒科)。
一项研究表明有两个大的分支:一个包括杯状病毒科、黄症病毒科和小RNA病毒科,另一个包括Alphatetraviridae、双RNA病毒科和囊状噬菌体科、野田病毒科和Permutotretraviridae。[18]
3.3 反义链RNA病毒
这些病毒具有多种类型的基因组,范围从单个RNA分子到八个片段不等。尽管它们具有多样性,但它们可能起源于节肢动物,并从那里开始多样化。[19]
3.4 卫星病毒
卫星病毒是需要另一种病毒帮助才能完成生命周期的病毒,很多卫星病毒也是已知的。他们的分类尚未确定。已经提议用以下四个属归类感染植物的正义链单链RNA卫星病毒——Albetovirus属、Aumaivirus属、Papanivirus属和Virtovirus属。[20] 一个包括Macronovirus病毒属的科——Sarthroviridae——已经被提出用于归类感染节肢动物的正义链单链RNA卫星病毒。
4 第III组——dsRNA病毒编辑
这一组中有十二个科和许多未下设的属和种。[21]
- 混合病毒科(Amalgaviridae)
- 双核糖核酸病毒科
- 金色病毒科(Chrysoviridae)
- 囊状噬菌体科(Cystoviridae)
- 内源核糖核酸病毒科(Endornaviridae)
- Hypoviridae
- 巨大双分核糖核酸病毒科(Megabirnaviridae)
- 分体病毒科(Partitiviridae)
- 小RNA病毒科(Picobirnaviridae)
- 呼肠孤病毒科——包括轮状病毒
- 整体病毒科(Totiviridae)
- Quadriviridae
- Botybirnavirus
- 未下设的物种
- Botrytis porri RNA virus 1
- Circulifer tenellus virus 1
- Colletotrichum camelliae filamentous virus 1
- Cucurbit yellows associated virus
- Sclerotinia sclerotiorum debilitation-associated virus
- Spissistilus festinus virus 1
5 第四组——正义链ssRNA病毒编辑
在这个群体中有3个目和34个科。此外,还有许多未下设的种和属。
- 网巢病毒目(Nidovirales)
- 动脉炎病毒科(Arteriviridae)
- 冠状病毒科(Coronaviridae)
- 海洋病毒科(Mesoniviridae)
- 杆状套病毒科(Roniviridae)
- 小RNA目(Picornavirales)
- 双顺反子科(Dicistroviridae)
- 传染性软化病毒科(Iflaviridae)
- 海洋RNA病毒科(Marnaviridae)
- 小RNA病毒科(Picornaviridae),包括脊髓灰质炎病毒、鼻病毒(一种普通感冒病毒)、甲型肝炎病毒
- 伴生班豆病毒科(Secoviridae),包括豇豆花叶病毒亚科(Comoviridae)
- Bacillariornavirus
- Dicipivirus
- Labyrnavirus
- Sequiviridae
- Kelp fly virus
- 芜菁黄花叶病毒目(Tymovirales)
- 甲型线形病毒科(Alphaflexiviridae)
- 乙型线形病毒科(Betaflexiviridae)
- 丙型线形病毒科(Gammaflexiviridae)
- 芜菁发黄镶嵌病毒科(Tymoviridae)
- 未设置目
- Alphatetraviridae
- Alvernaviridae
- 星状病毒科(Astroviridae)
- 杆菌状核糖核酸病毒科(Barnaviridae)
- 甜菜坏死黄脉病毒科Benyviridae
- 雀麦花叶病毒科(Bromoviridae)
- 杯状病毒科(Caliciviridae)——包括诺沃克病毒
- Carmotetraviridae
- 黄化丝状病毒科(Closteroviridae)
- 黄病毒科(Flaviviridae)——包括黄热病病毒、西尼罗病毒、丙型肝炎病毒、登革热病毒、寨卡病毒
- 肝炎病毒科(Hepeviridae)
- 低毒性病毒科(Hypoviridae)
- 光滑病毒科(Leviviridae)
- 黄症病毒科(Luteoviridae)——包括大麦黄矮病毒
- Polycipiviridae
- 裸露核糖核酸病毒(Narnaviridae)
- 野田病毒科('Nodaviridae')
- Permutotetraviridae
- 马铃薯Y病毒科(Potyviridae)
- Sarthroviridae
- Statovirus
- 多哥病毒科——包括风疹病毒、罗斯河病毒、辛德比斯病毒、基孔肯雅病毒
- Tombusviridae
- Virgaviridae[21]
- 未设置的属
- Blunervirus
- Cilevirus
- Higrevirus
- 悬钩子病毒属(Idaeovirus)
- Negevirus
- 欧尔密病毒属(Ourmiavirus)
- Polemovirus
- Sinaivirus
- 南方菜豆花叶病毒属(Sobemovirus)
- 未下设的种
- Acyrthosiphon pisum virus
- Bastrovirus
- Blackford virus
- Blueberry necrotic ring blotch virus
- Cadicistrovirus
- Chara australis virus
- Extra small virus
- Goji berry chlorosis virus
- Hepelivirus
- Jingmen tick virus
- Le Blanc virus
- Nedicistrovirus
- Nesidiocoris tenuis virus 1
- Niflavirus
- Nylanderia fulva virus 1
- Orsay virus
- Osedax japonicus RNA virus 1
- Picalivirus
- Plasmopara halstedii virus
- Rosellinia necatrix fusarivirus 1
- Santeuil virus
- Secalivirus
- Solenopsis invicta virus 3
- Wuhan large pig roundworm virus
卫星病毒
- Sarthroviridae
- Albetovirus
- Aumaivirus
- Papanivirus
- Virtovirus
- Chronic bee paralysis virus[22]
6 第五组——负义sRNA病毒编辑
除了丁型肝炎病毒之外,这一组病毒被归入一个单一的门——阴性病毒门(Negarnaviricota)。这个门被分成两个亚门——Haploviricotina和Polyploviricotina。在Haploviricotina亚门,目前公认有4个纲:春秋病毒纲、Milneviricetes、蒙吉病毒纲和云长病毒纲。在Polyploviricotina亚门中,公认有2个纲:Ellioviricetes和Insthoviricetes。
这个群体目前承认6个纲、7个目和24个科。一些种和属未下设。[23]
- Phylum Negarnaviricota[23]
- Subphylum Haploviricotina
- Class Chunqiuviricetes
- Muvirales
- Qinviridae
- Muvirales
- Class Milneviricetes
- Serpentovirales
- Aspiviridae
- Serpentovirales
- Class Monjiviricetes
- Jingchuvirales
- Chuviridae
- 单股反链病毒目(Mononegavirales)
- 玻那病毒科(Bornaviridae),玻纳疾病病毒(Borna disease virus)
- 纤维病毒科(Filoviridae),包括埃波拉病毒, 马尔堡病毒
- Mymonaviridae
- Nyamiviridae[24]
- 副黏液病毒科(Paramyxoviridae),包括Measles virus, Mumps virus, Nipah virus, Hendra virus, and NDV
- Pneumoviridae — 包括RSV and Metapneumovirus
- 弹状病毒科(Rhabdoviridae),包括狂犬病
- Sunviridae
- Genus Anphevirus
- Genus Arlivirus
- Genus Chengtivirus
- Genus Crustavirus
- Genus Wastrivirus
- Jingchuvirales
- Class Yunchangviricetes
- Goujianvirales
- Yueviridae
- Goujianvirales
- Class Chunqiuviricetes
- Subphylum Polyploviricotina
- Class Ellioviricetes
- Bunyavirales
- 沙状病毒科(Arenaviridae)包括塞拉热病毒
- Cruliviridae
- Feraviridae
- Fimoviridae
- Hantaviridae
- Jonviridae
- Nairoviridae
- 本雅病毒科(Bunyaviridae)
- Phasmaviridae
- Phenuiviridae
- Tospoviridae
- Genus Tilapineviridae
- Bunyavirales
- Class Insthoviricetes
- Articulavirales
- Amnoonviridae — 包括Taastrup virus
- 正黏液病毒科(Orthomyxoviridae)— 包括Influenza viruses
- Articulavirales
- Class Ellioviricetes
- Subphylum Haploviricotina
- 以下未设属:
- Genus Deltavirus — 包括丁型肝炎病毒(Hepatitis D virus)
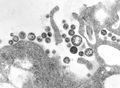
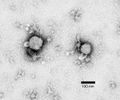
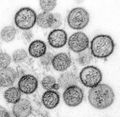
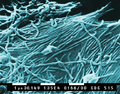
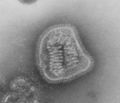
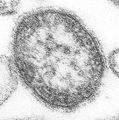
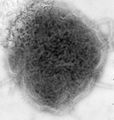
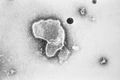
7 图片展示编辑
拉萨热病毒(沙状病毒科)
淋巴细胞性脉络丛脑膜炎病毒(沙状病毒科)
汉坦病毒(本雅病毒科)
马尔堡病毒(丝状病毒科)
埃博拉病毒(丝状病毒科)
流感(正粘病毒科)
麻疹(副粘病毒科)
腮腺炎病毒(副粘病毒科)
人呼吸道合胞病毒(副粘病毒科)
副流感病毒(副粘病毒科)
狂犬病病毒(弹状病毒科)
水泡性口炎病毒(弹状病毒科)
参考文献
- [1]
^MeSH, retrieved on 12 April 2008..
- [2]
^"Listing in Taxonomic Order—Index to ICTV Species Lists". Retrieved 2008-04-11..
- [3]
^Drake JW, Holland JJ (November 1999). "Mutation rates among RNA viruses". Proc. Natl. Acad. Sci. U.S.A. 96 (24): 13910–3. doi:10.1073/pnas.96.24.13910. PMC 24164. PMID 10570172..
- [4]
^Nguyen M, Haenni AL (2003). "Expression strategies of ambisense viruses". Virus Res. 93 (2): 141–50. doi:10.1016/S0168-1702(03)00094-7. PMID 12782362..
- [5]
^Patton JT (editor). (2008). Segmented Double-stranded RNA Viruses: Structure and Molecular Biology. Caister Academic Press. ISBN 978-1-904455-21-9..
- [6]
^Steinhauer DA, Holland JJ (1987). "Rapid evolution of RNA viruses". Annu. Rev. Microbiol. 41: 409–33. doi:10.1146/annurev.mi.41.100187.002205. PMID 3318675..
- [7]
^Boutwell CL, Rolland MM, Herbeck JT, Mullins JI, Allen TM (October 2010). "Viral Evolution and Escape during Acute HIV-1 Infection". J. Infect. Dis. 202 (Suppl 2): S309–14. doi:10.1086/655653. PMC 2945609. PMID 20846038..
- [8]
^Bukh J, Purcell RH, Miller RH (August 1994). "Sequence analysis of the core gene of 14 hepatitis C virus genotypes". Proc. Natl. Acad. Sci. U.S.A. 91 (17): 8239–43. doi:10.1073/pnas.91.17.8239. PMC 44581. PMID 8058787..
- [9]
^Tuplin A, Evans DJ, Simmonds P (October 2004). "Detailed mapping of RNA secondary structures in core and NS5B-encoding region sequences of hepatitis C virus by RNase cleavage and novel bioinformatic prediction methods". J. Gen. Virol. 85 (Pt 10): 3037–47. doi:10.1099/vir.0.80141-0. PMID 15448367..
- [10]
^Wolf YI, Kazlauskas D, Iranzo J, Lucía-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Koonin EV (2018) Origins and Evolution of the Global RNA Virome. MBio 9(6) pii: e02329-18.
- [11]
^Francki, R.I.B; Fauquet, C. M.; Knudson, D. L.; Brown, F. (1991). Classification and nomenclature of viruses. Fifth report of the International Committee on Taxonomy of Viruses, Archives of Virology (Suppl. 2). ISBN 978-3-7091-9163-7..
- [12]
^Koonin EV (September 1991). "The phylogeny of RNA-dependent RNA polymerases of positive-strand RNA viruses". J. Gen. Virol. 72 (Pt 9): 2197–206. doi:10.1099/0022-1317-72-9-2197. PMID 1895057..
- [13]
^Rozanov MN, Koonin EV, Gorbalenya AE (August 1992). "Conservation of the putative methyltransferase domain: a hallmark of the 'Sindbis-like' supergroup of positive-strand RNA viruses". J. Gen. Virol. 73 (Pt 8): 2129–34. CiteSeerX 10.1.1.532.7367. doi:10.1099/0022-1317-73-8-2129. PMID 1645151..
- [14]
^Koonin EV, Dolja VV (1993). "Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences". Crit. Rev. Biochem. Mol. Biol. 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709..
- [15]
^Ward CW (1993). "Progress towards a higher taxonomy of viruses". Res Virol. 144 (6): 419–453. doi:10.1016/S0923-2516(06)80059-2. PMID 8140287..
- [16]
^Koonin EV, Wolf YI, Nagasaki K, Dolja VV (December 2008). "The Big Bang of picorna-like virus evolution antedates the radiation of eukaryotic supergroups". Nat. Rev. Microbiol. 6 (12): 925–39. doi:10.1038/nrmicro2030. PMID 18997823..
- [17]
^Ghabrial SA (1998) Origin, adaptation and evolutionary pathways of fungal viruses. Virus Genes 16(1):119-131.
- [18]
^Gibrat JF, Mariadassou M, Boudinot P, Delmas B (2013). "Analyses of the radiation of birnaviruses from diverse host phyla and of their evolutionary affinities with other double-stranded RNA and positive strand RNA viruses using robust structure-based multiple sequence alignments and advanced phylogenetic methods". BMC Evol. Biol. 13: 154. doi:10.1186/1471-2148-13-154. PMC 3724706. PMID 23865988..
- [19]
^Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, Qin XC, Xu J, Holmes EC, Zhang YZ (2015) Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses. Elife 4. doi: 10.7554/eLife.05378..
- [20]
^Krupovic M, Kuhn JH, Fischer MG (2016) A classification system for virophages and satellite viruses. Arch Virol 161(1):233-247 doi: 10.1007/s00705-015-2622-9.
- [21]
^Adams MJ, Antoniw JF, Kreuze J (2009). "Virgaviridae: a new family of rod-shaped plant viruses". Arch Virol. 154 (12): 1967–72. doi:10.1007/s00705-009-0506-6. PMID 19862474..
- [22]
^Pankovics P, Boros Á, Kiss T, Engelmann P, Reuter G (2019) Genetically highly divergent RNA virus with astrovirus-like (5'-end) and hepevirus-like (3'-end) genome organization in carnivorous birds, European roller (Coracias garrulus). Infect Genet Evol.
- [23]
^Klein, Donald W.; Prescott, Lansing M.; Harley, John (1993). Microbiology. Dubuque, Iowa: Wm. C. Brown. ISBN 978-0-697-01372-9..
- [24]
^Mihindukulasuriya, K. A.; Nguyen, N. L.; Wu, G.; Huang, H. V.; Travassos da Rosa, A. P.; Popov, V. L.; Tesh, R. B.; Wang, D. (2009). "Nyamanini and Midway viruses define a novel taxon of RNA viruses in the order Mononegavirales". J. Virol. 83 (10): 5109–16. doi:10.1128/JVI.02667-08. PMC 2682064. PMID 19279111..
- [25]
^Kondo, H.; Chiba, S.; Toyoda, K.; Suzuki, N. (2012). "Evidence for negative-strand RNA virus infection in fungi". Virology. 435 (2): 201–9. doi:10.1016/j.virol.2012.10.002. PMID 23099204..
暂无