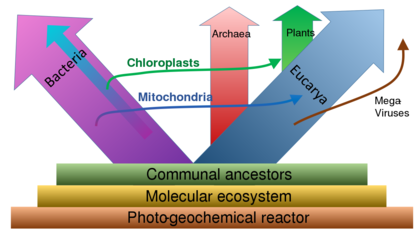
真核生物
编辑真核生物是细胞的细胞核被膜包围的生物,而原核生物(细菌和古细菌)没有膜结合细胞器。真核生物属于真核生物界(Eukaryota)或真核生物域(Eukarya)的分类阶元。Eukaryota 和Eukarya的名字来自希腊的εὖ(eu,“好”或“真”)和κάρυον(karyon,“坚果”或“内核”)[6].真核细胞还含有其他膜结合细胞器,如线粒体和高尔基体,此外,一些植物和藻类细胞含有叶绿体。与单细胞古菌和细菌不同,真核生物也可以是多细胞生物有机体,包括由许多细胞类型组成的有机体,形成不同种类的组织。动物和植物是最熟悉的真核生物。
真核生物可以通过有丝分裂进行无性繁殖,也可以通过减数分裂和配子融合进行有性繁殖。在有丝分裂中,一个细胞分裂产生两个基因相同的细胞。在减数分裂中,脱氧核糖核酸复制之后是两轮细胞分裂,产生四个单倍体子细胞。这些细胞充当性细胞(配子)。每个配子只有一套染色体,每一套都是减数分裂过程中遗传重组产生的相应亲本染色体对的独特组合。
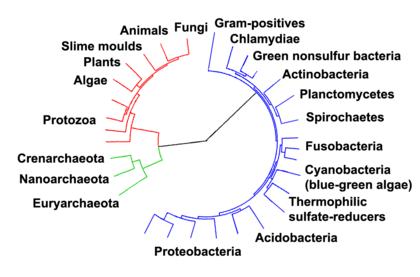
真核生物分类阶元似乎是单系的,并且构成了三域系统中的分类阶元之一。另外两个分类阶元,细菌和古细菌,是没有上述特征的原核生物。真核生物代表的只是所有生物中的一小部分。[1]然而,由于真核生物的体积通常要大得多,因此它们在世界范围内的总生物量估计约等于原核生物的生物量。[1]真核生物大约在16-21亿年前的元古代进化。
目录编辑
1 历史编辑
真核生物的概念被认为是法国生物学家爱德华·查顿(1883-1947)提出的。加拿大微生物学家罗杰·斯坦尼尔和荷兰裔美国微生物学家范尼尔于1962年决定性地重新引入了原核生物和真核生物的术语。查顿在他1937年的著作《Titres et Travaux Scientifiques》[2]中提出了这两个术语,称细菌为原核生物,细胞中有细胞核的生物体为真核生物。然而,他只在一段中提到了这一点,这个想法实际上被忽略了,直到斯坦尼尔和范尼尔重新发现了查顿的陈述。[3]
在1905年和1910年,俄罗斯生物学家康斯坦丁·梅雷施科夫斯基(1855-1921)认为质体是与非光合(异养)宿主共生的还原型蓝藻,该宿主本身是由变形虫样宿主和形成细胞核的细菌样细胞共生形成的。因此,植物遗传了蓝藻的光合作用的功能。[4]
1967年,琳·马古利斯在她的论文《有丝分裂细胞的起源》中为真核细胞叶绿体和线粒体的起源——内共生提供了微生物学证据。[5]20世纪70年代,卡尔·沃尔斯探索微生物系统发育,研究16S核糖体核糖核酸的变异。卡尔·沃尔斯的工作有助于揭示真核生物的起源和两个重要真核生物细胞器,线粒体和叶绿体的共生。1977年,沃尔斯和乔治·福克斯引入了“第三种生命形式”的概念,他们称之为古细菌(Archaebacteria);1990年,·沃尔斯、奥托·坎德勒和马克·威尔利斯把它重新命名为古菌(Archaea)。[3]
1979年,古尔德(G. W. Gould)和德里宁(G. J. Dring)提出真核细胞的细胞核来自革兰氏阳性菌形成内生孢子的能力。在1987年和后来的论文中,托马斯·卡瓦利-史密斯提出细胞核和内质网的膜首先是通过原核生物的质膜内陷形成的。20世纪90年代,其他几位生物学家提出了细胞核的内共生起源学说,有效地复兴了梅雷施科夫斯基的理论。[4]
2 细胞特征编辑
真核细胞通常比原核细胞大得多,其体积约为原核细胞的10,000倍。[6]它们有多种内部膜结合结构,称为细胞器,以及由微管、微丝和中间丝组成的细胞骨架,在确定细胞的组织和形状方面起着重要作用。真核生物的脱氧核糖核酸被分成几个称为染色体的线性束,它们在核分裂过程中被微管纺锤体分开。
2.1 内膜
真核细胞包括多种膜结合结构,统称为内膜系统。[7]叫做囊泡和液泡的简单隔室,可以通过出芽的方式从其他膜上形成。许多细胞通过内吞过程摄取食物和其他物质,在内吞过程中,外膜内陷,然后收缩形成囊泡。[8]很可能大多数其他膜结合细胞器最终都来源于这种囊泡。或者,细胞产生的一些产物可以通过胞吐作用留在囊泡中。
细胞核被双层膜(通常称为核膜或核被膜)包围,其孔隙允许物质进出。[9]核膜的各种管状和片状延伸形成内质网,参与蛋白质运输和成熟过程。内质网包括粗面内质网,核糖体附着在那里合成可以进入内部空间或内腔的蛋白质。随后,它们通常进入囊泡,囊泡从光面内质网萌芽。[10]在大多数真核生物中,这些携带蛋白质的囊泡在堆叠起来的高尔基体扁平囊泡(池)中被释放和进一步修饰。[11]
囊泡可以专门用于各种目的。例如,溶酶体含有分解细胞质中大多数生物分子的消化酶。[12]过氧化物酶体用于分解过氧化物,否则过氧化物是有毒的。许多原生动物有可收缩的液泡和挤出物,用于收集和排出多余的水,还有外分泌体,用于转移捕食者或捕捉猎物的物质。在高等植物中,细胞的大部分体积被一个中央液泡占据,该液泡主要含有水,并主要维持细胞的渗透压。
2.2 线粒体和质体
线粒体是除了一种真核生物以外的所有真核生物中的细胞器。线粒体通过将糖转化为三磷酸腺苷为真核细胞提供能量。[13]它们周围有两层膜,每一层都是磷脂双层;其内部折叠成称为嵴的内陷,嵴是有氧呼吸进行的场所。
线粒体外膜是可自由渗透的,几乎允许任何物质进入膜间空间,而线粒体内膜是半透膜,因此只允许一些必需的物质进入线粒体基质。
线粒体含有它们自己的脱氧核糖核酸,该脱氧核糖核酸与细菌脱氧核糖核酸有着密切的结构相似性,并且编码在结构上更接近细菌RNA而不是真核生物RNA的rRNA和tRNA基因。[14]现在人们普遍认为它们是由内共生原核生物,可能是变形菌,发展而来的。
一些真核生物,如贾第鞭毛虫和毛滴虫等后滴门类,以及变形虫类佩罗米夏虫,似乎缺乏线粒体,但都被发现含有线粒体衍生的细胞器,如氢化酶体和纺锤剩体,因此二次失去了线粒体。它们通过对从环境中吸收的营养物的酶促作用获得能量。通过横向基因转移,后滴门类的类单鞭滴虫属(Monocercomonoides)的生物也获得了一个胞质硫动员系统,该系统提供蛋白质合成所需的铁和硫簇。正常的线粒体铁硫簇途径已经二次丢失。[15]
植物和各种藻类也有质体。质体也有自己的脱氧核糖核酸,由内共生菌发育而来,在这种情况下内共生菌是蓝藻。它们通常采取叶绿体的形式,像蓝细菌一样,包含叶绿素,通过光合作用产生有机化合物(如葡萄糖)。其他质体参与储存食物。虽然质体可能只有一个起源,但并非所有含质体的基团都是密切相关的。相反,一些真核生物是通过次级内共生或摄食从其他生物那里获得它们的。[16]光合细胞和叶绿体的捕获和隔离过程发生在许多现代真核生物中,被称为盗食质体(kleptoplasty)。
内共生起源学说还被用在了解释细胞核和真核生物的鞭毛起源上。[17]
2.3 细胞骨架结构
许多真核生物有细长的游动细胞质突起,称为鞭毛,或称为纤毛的类似的结构。鞭毛和纤毛有时被称为波动足,[18]并参与运动、进食和感觉等不同的生理功能。边贸和纤毛主要由微管蛋白组成。真核生物的鞭毛和纤毛与原核鞭毛完全不同。它们由中心粒产生的一束微管支撑,其特征是围绕两个单线态排列成九对。鞭毛也可能有茸毛(hairs),或称茸鞭(mastigonemes),以及连接细胞膜和内杆的鳞片。它们的内部与细胞的细胞质是连续的。
由肌动蛋白和肌动蛋白结合蛋白如α-肌动蛋白、纤维蛋白、细丝蛋白组成的微丝状结构也存在于膜下皮层和束中。微管的马达蛋白,例如动力蛋白或驱动蛋白和肌动蛋白,例如肌球蛋白,提供了鞭毛结构的网络的动态特性。
中心粒甚至经常出现在没有鞭毛的细胞和群体中,但是针叶树和开花植物都没有鞭毛。中心粒通常发生在易被诱发形成各种微管根的群体中。这些构成细胞骨架结构的主要组成部分,通常在各自的细胞分裂过程中组装,一个鞭毛保留在母体中,另一个来源于母体。中心粒在核分裂过程中产生纺锤体。[19]
细胞骨架结构的重要性在细胞形状的确定中得到了强调,它们也是迁移反应如趋化性和化学增活现象的重要组成部分。一些原生动物有各种微管支持的细胞器。其中包括放射虫和太阳虫。它们产生用于漂浮或捕捉猎物的轴足动物,以及粘着植物,它们有一种特殊的鞭毛状细胞器,称为附着鞭毛(haptonema)。
2.4 细胞壁
植物和藻类、真菌和大多数囊泡藻类细胞具有细胞壁,这是细胞膜外的一层结构,为细胞提供结构支持、保护和过滤机制。当水进入细胞时,细胞壁也防止过度膨胀。[20]
构成陆生植物初生细胞壁的主要多糖是纤维素、半纤维素和果胶。纤维素微纤维通过半纤维素系绳连接形成纤维素-半纤维素网络,该网络嵌入果胶基质中。初级细胞壁中最常见的半纤维素是木葡聚糖。[21]
3 真核细胞之间的差异编辑
尽管动物和植物是最熟悉的真核生物,但是真核细胞有许多不同类型,因此为理解真核生物结构提供了一个极好的起点。然而,真菌和许多原生生物有一些实质性的区别。
3.1 动物细胞
所有动物都是真核生物。动物细胞不同于其他真核生物的细胞,尤其是植物细胞,因为动物细胞没有细胞壁和叶绿体,液泡较小。由于缺乏细胞壁,动物细胞可以转变成多种形状。吞噬细胞甚至可以吞噬其他细胞结构。
3.2 植物细胞
植物细胞与其他真核生物的细胞差异很大。它们具有以下显著特点:
- 一个大的中央液泡(被称为液泡膜的膜结构包围),它维持细胞的渗透压并控制细胞质和细胞液之间的分子运动。[22]
- 由原生质体沉积在细胞膜外部的含有纤维素、半纤维素和果胶的初级细胞壁;这与含有几丁质的真菌细胞壁和主要由肽聚糖构成的原核生物的细胞壁形成了对比。
- 胞间连丝,是细胞壁中连接相邻细胞并允许植物细胞与相邻细胞进行通讯的孔。[23]动物在相邻细胞之间有不同形式但功能相似的缝隙连接系统。
- 质体,尤其是叶绿体,是含有叶绿素的细胞器,叶绿素是赋予植物绿色并使其进行光合作用的色素。
- 苔藓植物和无核维管植物精子细胞中只有鞭毛和中心粒。精子苏铁s和银杏是大型复杂的细胞,有成百上千的鞭毛游动。
- 苔藓植物和无种子维管植物在精细胞中只有鞭毛和中心粒。[24]苏铁和银杏的精子是大型复杂的细胞,游动时有成百上千个鞭毛同时参与工作。[25]
- 针叶树(松柏门,Pinophyta)和开花植物(被子植物,Angiospermae)的细胞缺少动物细胞中的鞭毛和中心粒。
3.3 真菌细胞
真菌的细胞与动物细胞最相似,只有以下三个特征除外:[26]
- 真菌具有含有几丁质的细胞壁
- 细胞间分隔较少;高等真菌的菌丝有叫做隔膜的多孔分隔物,它允许细胞质、细胞器,有时还有细胞核通过。原始真菌几乎没有隔膜,所以每一个有机体本质上都是一个巨大的多核超细胞;这些真菌被描述为多核细胞。
- 只有最原始的真菌,壶菌,才有鞭毛。
3.4 其他真核细胞
一些真核生物群体有独特的细胞器,例如灰藻细胞中的共生体(不寻常的叶绿体),[27]黏着藻类细胞的附着鞭毛,或隐藻的喷射体。其他结构,如伪足,在不同的真核生物群中以不同的形式被发现,如裂片变形虫或网状有孔虫。[28]
4 生殖编辑
细胞分裂通常通过有丝分裂进行无性繁殖,这一过程允许每个子核接受每个染色体的一个拷贝。大多数真核生物也有一个涉及有性生殖的生命周期,在单倍体阶段和二倍体阶段交替进行,单倍体阶段每个细胞中只有一个染色体拷贝,二倍体阶段每个细胞中有两个染色体拷贝。二倍体阶段由两个单倍体配子融合形成合子,合子可以通过有丝分裂分裂或通过减数分裂减少染色体。这种模式在不同物种间有很大的不同。动物没有多细胞单倍体阶段,但是每一代植物可以由单倍体和二倍体多细胞阶段组成。
真核生物比原核生物的表面积与体积之比更小,因此代谢率更低,世代时间更长。[29]
有性生殖的进化可能是真核生物的原始和基本特征。基于系统发育分析,达克和罗杰提出兼性性行为存在于所有真核生物的共同祖先中。[30]阴道毛滴虫和肠贾第鞭毛虫都有一组在减数分裂中起作用的核心基因,而这两种生物体以前被认为是进行无性生殖的。[31][32]由于这两个物种是早期从真核进化树中分化出来的谱系的后代,因此可以推断所有真核生物的共同祖先中可能存在核心减数分裂基因,因此也可能存在性别。[31][32]曾经被认为是进行无性生殖的真核物种,例如利什曼原虫属的寄生原生动物,已经被证明有一个有性生殖周期。[33]此外,现在有证据表明阿米巴原虫,以前被认为是进行无性生殖的,在古代是节能型有性生殖的生物,现在大多数无性生殖群体可能是最近独立出现的。[34]
5 分类编辑
在古代,动物和植物的两个谱系是公认的。林奈赋予它们“界(Kingdom)”的分类等级。虽然他把真菌和植物包括在内,但后来人们意识到它们非常不同,应该有一个独立的界的分类去描述真菌,直到20世纪80年代,这个界的组成才完全清楚。[35]当各种单细胞真核生物被发现时,它们最初与植物或动物放在一起。1818年,德国生物学家乔治·戈德福斯创造了原生动物一词,用来指代纤毛虫等生物,[36]这个群体一直扩大到包括所有单细胞真核生物,并于1866年被艾伦斯特·赫克尔赋予了自己界的名称——原生动物。[37][38]因此,真核生物由四个王国组成:
- 原生生物界
- 植物界
- 真菌界
- 动物界
原生生物被理解为“原始形式”,因此是一个进化的等级,由它们原始的单细胞性质统一在一起。[38]生命树深层分裂的谜底揭示过程真正始于脱氧核糖核酸测序,使卡尔·乌斯提出的阶元(domain)而不是界(kingdom)作为最高等级的系统,将所有真核生物王国合并到真核生物阶元之下。[39]与此同时,原生动物建树的工作得到了加强,至今仍在积极进行。尽管在原生动物分类这一领域没有达成共识,但是已经提出了几个替代分类方法。
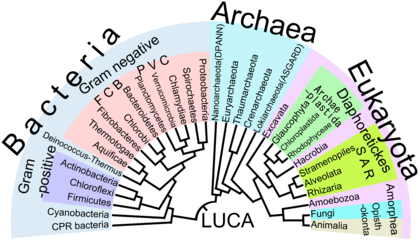
真核生物是一个分支,通常被认为是古细菌Asgard群类中海姆达古菌门(Heimdallarchaeota)的姐妹。[39]基本分类群组是Opimoda, Diphoda, Discoba和 Loukozoa。真核生物的根通常被认为为接近甚至是属于Discoba群组。
2005年为国际原生生物学家协会(International Society of Protistologists)编制的分类反映了当时分类学家的共识,[40]将真核生物分成六个假定为是单系的“超群”。然而,在同一年(2005年),有人对其中一些超群,特别是对囊泡藻超群,是否是单系化的表示怀疑,[41]2006年的一项回顾调查中指出,在假定的六个超群中,有几个缺乏证据。[42]2012年修订的分类[43]承认五个超群。
| 古生菌(或原生动物) | 陆生植物、绿藻、红藻和灰胞藻 |
| SAR超群 | 不等鞭毛类(褐藻、硅藻等)、囊泡虫类和有孔虫类(有孔虫、放射虫和各种其他变形虫原生动物)。 |
| 古虫界 | 各种鞭毛原生动物 |
| 变形虫界 | 大多数球形变形虫和黏菌 |
| 后鞭毛生物界 | 动物、真菌、领鞭虫类等。 |
也存在较小的真核生物群类,[44]它们——特别是定鞭藻门(Haptophyta)、隐藻门(Cryptophyta)、中阳虫门(Centrohelida)、末丝虫门(Telonemia)、微微藻门(Picozoa)[45]、无足滴虫门(Apusomonadida)、锚单胞虫门(Ancyromonadida)、早别虫门(Breviatea)和胶网藻(Collodictyon),[46]这些小类群的位置不确定或似乎不属于主要群体[54]。总的来说,尽管取得了进展,但真核生物的进化史和分类仍然存在非常大的不确定性。正如罗杰和辛普森在2009年所说,“随着我们对真核生物生命树理解的当前变化速度,我们应该谨慎行事。”[47]
在2016年4月发表于《自然微生物学》的一篇文章中,作者们“再次强调,我们周围看到的生命——植物、动物、人类和其他所谓的真核生物——只占世界生物多样性的一小部分。”[48]他们“基于信息系统的遗传,而不是脂质或其他细胞结构”,将真核生物分类。加州大学伯克利分校的吉利安·班菲尔德(Jillian F. Banfield)和其他科学家使用一台超级计算机,根据3000个物种的脱氧核糖核酸(包括2072个已知物种和1011个新报道的微生物)生成了一个新的生命树图表,他们从不同的环境中收集了这些微生物的脱氧核糖核酸。[49][49]随着测序能力增强,班菲尔德和他的团队能够进行宏基因组测序——“在同一时间内对整个生物群落进行测序,并仅根据它们的基因来挑选个体群体。”[48]
5.1 系统演化
在20世纪80年代和90年代构建的rRNA树将大多数真核生物留在一个未解决的“冠”群中(从技术上讲不是真正的冠),该组通常由线粒体嵴的形式划分。少数缺乏线粒体的群体单独地分支,因此它们被认为是原始的;但是现在这被认为是一个长枝发展方向,并且这类群体已经被认为是第二次失去了线粒体。[50][51]
截至2011年,人们普遍认为,有孔虫类(Rhizaria)、不等鞭毛类(Stramenopiles)和囊泡虫类(Alveolata)属于一个被称为SAR超类群的分支中的,因此有孔虫类不是主要的真核生物群之一;变形虫类(Amoebozoa)和后鞭毛类(Opisthokonta)都是单系的,并形成一个分支,通常称为单鞭毛生物(unikonts)。[52][53][54][55]除此之外,其他分类似乎没有达成共并识。
据估计,真核生物可能有75个不同的世系。[56]这些世系大多是原生生物。
已知的真核生物基因组大小从牛巴贝斯虫(Babesia bovis)的8.2 Mb到海洋原甲藻(Prorocentrum micans)的112,000–220,050 Mb不等,表明祖先真核生物的基因组在其进化过程中经历了相当大的变化。[56]所有真核生物的最后一个共同祖先被认为是吞噬性原生生物,具有细胞核、至少一个中心粒和纤毛、兼性需氧线粒体、有性生殖(减数分裂和配子融合),一个具有细胞壁的休眠囊,其细胞壁由几丁质和/或纤维素和过氧化物酶体组成。[56]后来的内共生导致质体在某些世系中的传播。
五个超群
Adl等人(2012年)和Burki(2014/2016年)在系统发育证据(特别是系统发育学)、[43]稀有基因组特征和形态学特征的共识基础上,提出了真核生物的全球树,其中隐生植物门(Cryptophyta)和微微藻门(picozoa)已在泛植物界中出现。[44][57][58][59][60][61]也有类似的包含了灰藻门(Glaucophyta)、隐藻门(Cryptista)(以及不同寻常的定鞭藻门(Haptista))的分类群被确立。
| Eukaryotes |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
在一些分析中,哈氏藻群(定鞭藻门+隐藻门)被放在的泛植物界附近,[52]但在其他分析中,它被嵌套在泛植物界内部。然而,最近的几项研究得出结论,定鞭藻门和隐藻门不形成一个单系群。[62]前者可以是SAR超类群的姐妹群类,后者是与泛植物界(广义植物)聚类到一起的类群。[63]
早些时候已经提出了将真核生物分为两个初级分支,分别来源于一个双鞭生物祖先的双鞭毛生物(泛植物界+SAR超类群+古虫界)和来源于单鞭毛生物祖先的单鞭毛生物(变形虫界和后鞭毛生物)。[64][65]2012年的一项研究得出了类似的划分,尽管人们注意到“单鞭毛生物”和“双鞭毛生物”这两个术语并没有在最初的意义上使用。[45]
Derelle等人(2015年)、Ren等人(2016年)、Yang等人(2017年)和Cavalier-Smith (2015年)的研究中出现了一组高度聚合和一致的树,包括其补充信息也相当一致,从而产生了一个更加保守和统一的树。它结合了卡瓦利-史密斯对基础Opimoda.的一些研究结果。[66][67][68][69][60][70]剩下仍然存在的主要争议是关于红藻门和双鞭毛动物根足亚界、定鞭界、隐藻亚界、微微藻界和末丝虫界的根和确切位置,上述生物其中许多种类可能是内共生的真核生物,即真核生物的内共生杂交产物。[71]泛植物界可能通过原核祖先的内共生获得叶绿体,该原核祖先与目前现存的蓝细菌——Gloeomargarita lithophora有关。[72][73][71]
| Eukaryotes |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Cavalier-Smith的树
托马斯·卡瓦里尔-史密斯(2010年, 2013年,2014年, 2017年和2018年[74]将真核生物进化树的根置于古虫类(Excavata)(腹侧进食槽由微管根支撑)和无槽眼虫门(grooveless Euglenozoa)和单系色藻界(Chromista)之间,它的发生与捕获红藻的单一内共生事件相关。He等人[75]特别支持将真核树生根在单系的盘嵴亚界(盘嵴总门(disciristata)+雅各巴虫界(Jakobida))和Amorphea-多貌总界(Diaphoretickes)之间。
| Eukaryotes |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
6 真核生物的起源编辑
6.1 化石
真核细胞的起源是生命进化的里程碑,因为真核生物包括所有复杂细胞和几乎所有多细胞生物。这一系列事件的时间很难确定;Knoll (2006)认为它们大约是在16-21亿年前发展起来的。一些疑源类至少在16.5亿年前就已为人所知,而可能的卷曲藻早在21亿年前就被发现了。[76]在22亿年前的古土壤中发现了类似地管衣属的真菌化石Diskagma。[77]
在加蓬古元古代法国B形成层的黑色页岩中发现了有组织的生物结构,年代为21亿年。真核生物当时可能已经进化了。[78]与现代群类明显相关的化石开始以红藻的形式出现在大约12亿年前,尽管最近的研究表明宾陀山盆地存在丝状藻类化石,时间可能可以追溯到16到17亿年前。[79]
生物标记表明至少干细胞真核生物出现得更早。澳大利亚页岩中甾烷的存在表明这些岩石中存在真核生物,年代为27亿年,[80][81]尽管有人认为该现象可能源于样品污染。[82]
无论何时起源,真核生物可能直到很久以后才成为生态优势;8亿年前海洋沉积物中锌含量的大幅上升归因于大量真核生物的增加,这些真核生物相对于原核生物更倾向于消耗和吸收锌。[83]
2019年4月,生物学家报告称,非常大的水母病毒或其亲属,可能是复杂真核细胞从简单原核细胞进化出的部分原因。[84]
6.2 与古细菌的关系
真核生物的核脱氧核糖核酸和遗传机制更像古细菌而不是细菌,这导致了一个有争议的建议,即真核生物应该与古细菌在新村(Neomura)枝中分为一组。在其他方面,如膜组成,真核生物类似于细菌。对此提出了三种主要解释:
- 真核生物是由两个或多个细胞完全融合而成的,其中细胞质由真细菌形成,[85]细胞核来自古细菌、病毒[86][87]或前细胞。[88][89]
- 真核生物从古细菌发展而来,并通过真细菌来源的原线粒体的内共生获得其真细菌特征。[90]
- 真核生物和古细菌是从改良的真细菌中分离出来的。
备选提案包括:
- 慢性细胞假说假设原始真核细胞是由古细菌和细菌的内共生作用形成的,第三种细胞称为慢性细胞。[91]
- 当前生命之树的普遍共同祖先(UCA)是一个复杂的有机体,它在大规模灭绝事件中幸存下来,而不是生命进化的早期阶段。真核生物,特别是无核细胞(细菌和古细菌)是通过还原缺失进化而来的,[92]因此真核生物的相似性源于原始特征的差异保留。
假设没有其他类群参与,细菌、古细菌和真核生物有三种可能的系统发育,其中每一种都是单系的。这些在下表中标记为1至3。始新世假说是对假说2的修正,在假说2中古细菌属于侧系群。(该表和假设的名称设定是基于哈里什和库尔兰,2017年的研究。[93])
| 1–两个帝国的假说 | 2–三个阶元域假说 | 3–Gupta的学说 | 4–始新世假说 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|
|
近年来,大多数研究者支持三个阶元域(3D)或始新世假说。rRNA分析支持始新世假说,显然该假说中真核生物的根在古虫界中。[76][94][94][94][94]一个支持始新世假说的分支图是:[94][94][94]
| Proteoarchaeota |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||
在这种情况下,Asgard群类被视为TACK群类的姐妹分类单元,它包括泉古菌门(Crenarchaeota,以前命名为eocytes)、奇妙的古菌——奇古菌(Thaumarchaeota)和其他门类。
2017年,这种学说遭到了强烈反对,认为真核生物没有出现在古细菌中。Cunha等人提出的分析支持三个阶元域(3D)或Woese假说(上表中的2个),拒绝始新世假说(上表中的第4个)。[94]基于对蛋白质结构域编码序列的分析,哈里什和库尔兰发现了对前两个帝国(2D)或迈尔假说(上表1)的有力支持。他们拒绝了始新世假说,认为可能性最小。[95][93]对他们分析的一种可能解释是,当前生命之树的普遍共同祖先(UCA)是一个经受住了进化瓶颈的复杂的有机体,而不是一个在生命历史早期出现的更简单的有机体。[92]
6.3 内膜系统和线粒体
内膜系统和线粒体的起源也不清楚。[96]吞噬作用假说提出,缺乏细胞壁的真核类型的膜首先随着内吞作用的发展而产生,而线粒体是作为内共生体通过摄入获得的。[97]共生假说提出,原真核生物依赖原线粒体获取食物,因此最终原真核生物生长到将原线粒体包围到体内。这里的细胞膜起源于线粒体被吞噬后,部分归因于线粒体基因(氢假说是一个特殊的版本)。[98]
Pisani等人(2007年)在一项利用基因组构建超树的研究中指出,除了证据表明从来没有无线粒体的真核生物外,还证明了真核生物是从与热原体目(Thermoplasmatales)密切相关的古菌和α-变形菌门之间的共生演化而来的,可能是由硫或氢驱动的共生现象。线粒体及其基因组是α-变形菌门内共生体的残余。[99]
6.4 假设
关于真核细胞是如何产生的,已经提出了不同的假设。这些假设可以分为两类——自体模型和嵌合体模型。
自体模型
自体模型表明,首先存在一个含有细胞核的原真核细胞,然后获得线粒体。[100]根据这个模型,一个大型原核生物在质膜中形成内陷,以便获得足够的表面积来维持细胞质体积。随着内陷在功能上的分化,一些成了独立的隔室——产生内膜系统,包括内质网、高尔基体、核膜和溶酶体等单层膜结构。[101]线粒体被认为来自需氧变形菌的内共生系统,并且假设所有没有获得线粒体的真核谱系都灭绝了。[102]叶绿体来自另一个涉及蓝细菌的共生事件。因为所有的真核生物都有线粒体,但不是所有的都有叶绿体,所以这一系列的内共生理论认为线粒体是第一位的。
嵌合体模型
嵌合体模型声称最初存在两个原核细胞——一个古细菌和一个细菌。这些生物的近亲似乎是Asgard古菌和(远亲) α-变形菌。[103][104]这些细胞经历了一个融合过程,通过物理融合或内共生,从而导致真核细胞的形成。在这些嵌合体模型中,一些研究进一步声称线粒体起源于细菌祖先,而另一些研究强调线粒体起源背后的内共生过程的作用。
互为表亲的科学家大卫和巴兹鲍姆提出的由内向外假说表明,自由生活的线粒体样细菌和古菌融合成一个原核细胞是在很长一段时间内逐渐发生的,而不是一口气吞噬。在这种情况下,古菌会用细胞突起捕捉需氧细菌,然后让它们存活下来,从它们身上吸取能量,而不是消化它们。在早期阶段,细菌仍然部分地与环境直接接触,古菌也不必为它们提供所有必需的营养。但最终古菌会完全吞噬细菌,在此过程中产生内部膜结构和核膜。[105]据推测,被称为嗜盐菌(halophiles)的太古代的群类经历了类似的过程,嗜盐菌从细菌中获得了多达1000个基因,远远超过了微生物世界中常见的常规水平基因转移,但这两种微生物在融合成一个真核细胞之前又分离了。[106]
基于共生过程,这些假说可以分为——系列共生理论(SET),[107][108][109]氢假说(主要是在不同物种之间发生氢转移的共生过程),[98]和共生假说。[110][111]
真核细胞是由两个原核生物之间的物理相互作用产生的假说的一个扩展版本是,真核生物的最后一个共同祖先是从参与合作关系的微生物群体或共体中获得基因组,以便在它们的环境中茁壮成长和生存。来自不同类型微生物的基因组会为了自身利益而相互补充,偶尔会导致它们之间的水平基因转移。有益基因的积累产生了真核细胞的基因组,其中包含了独立所需的所有基因。[112]
根据一系列内共生理论(由琳·马古利斯倡导),运动厌氧菌(如螺旋菌)和嗜热嗜酸菌泉古菌(如自然界中产生硫化物的热质菌)的结合产生了今天的真核生物。这种结合建立了一种能动的有机体,能够生活在已经存在的酸性和含硫的水中。众所周知,氧气会对缺乏所需代谢机制的生物体造成毒性。因此,古细菌为细菌提供了非常有益的还原环境(硫和硫酸盐被还原成硫化物)。在微需氧条件下,氧气被还原成水,从而创造了一个互利的平台。另一方面,细菌为古细菌提供了必需的发酵产物和电子受体以及它的运动特性,从而获得了有机体的游动运动。真核细胞的核基因组起源于细菌和古细菌的联合。螺旋体产生了真核细胞的运动特征。α-变形菌和蓝细菌祖先的共生统一分别导致线粒体和质体的起源。例如,已知枝硫菌属(Thiodendron)起源于一种外共生过程,该过程基于两种细菌——脱硫杆菌和螺旋体——之间存在的硫的相似同构。然而,这种基于能动共生的关联从未在实际中观察到。也没有证据表明古生菌和螺旋体适应强烈的酸碱环境。[100]
在氢假说中,厌氧和自养产甲烷古细菌(宿主)与α变形菌(共生体)的共生联系产生了真核生物。宿主利用氢气(H2)和二氧化碳(CO2)在共生体能够有氧呼吸的同时产生甲烷,排出作为厌氧发酵过程副产物H2和CO2.。宿主的产甲烷环境充当了H2的沉淀池,导致细菌发酵加剧。内共生基因转移(EGT)是宿主获得共生体碳水化合物代谢并转变为异养型生物的催化剂。随后,宿主的产甲烷能力丧失。因此,异养细胞器(共生体)的起源与真核生物谱系的起源相同。在这个假设中,H2的存在代表了从原核生物中锻造出真核生物的选择性力量。
共生假说与氢假说形成对比,提出了两个共生事件的存在。根据这一理论,真核细胞的起源是基于产甲烷古细菌和δ变形菌之间的代谢共生。这种共养共生最初是由厌氧环境下不同物种之间的H2转移促成的。在早期阶段,α变形菌成为这种整合的成员,后来发展成线粒体。从δ变形菌到古细菌的基因转移导致产甲烷古细菌发育成细胞核。古细菌构成了遗传装置,而δ变形菌有助于细胞质特征的形成。这一理论结合了细胞核进化时的两种选择力——(a)代谢分配的存在,以避免合成代谢和分解代谢细胞途径共存的有害影响,以及(b)在获得线粒体并失去甲烷生成后,由于古细菌基因内含子的大量扩散而导致的异常蛋白质生物合成的预防。
6+系列内共生学说
Pitts和Galbanón提出了一个复杂的6+系列古生菌和细菌内共生事件的场景,其中线粒体和Asgard相关的古生菌在真核发育的后期获得,可能结合起来,作为次级内共生生物。[113][114]但是这个学说被认为是伪学说。[115]
参考文献
- [1]
^Whitman WB, Coleman DC, Wiebe WJ (June 1998). "Prokaryotes: the unseen majority" (PDF). Proceedings of the National Academy of Sciences of the United States of America. 95 (12): 6578–6583. Bibcode:1998PNAS...95.6578W. doi:10.1073/pnas.95.12.6578. PMC 33863. PMID 9618454..
- [2]
^Chatton, Édouard (1937). Titres Et Travaux Scientifiques (1906-1937) De Edouard Chatton. Sète: Impr. E. Sottano..
- [3]
^Sapp J (June 2005). "The prokaryote-eukaryote dichotomy: meanings and mythology". Microbiology and Molecular Biology Reviews. 69 (2): 292–305. doi:10.1128/MMBR.69.2.292-305.2005. PMC 1197417. PMID 15944457..
- [4]
^Martin WF, Garg S, Zimorski V (September 2015). "Endosymbiotic theories for eukaryote origin". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 370 (1678): 20140330. doi:10.1098/rstb.2014.0330. PMC 4571569. PMID 26323761..
- [5]
^Sagan L (March 1967). "On the origin of mitosing cells". Journal of Theoretical Biology. 14 (3): 255–274. doi:10.1016/0022-5193(67)90079-3. PMID 11541392..
- [6]
^Yamaguchi, Masashi; Worman, Cedric O'Driscoll (2014). "Deep-sea microorganisms and the origin of the eukaryotic cell" (PDF). Jpn. J. Protozool. 47 (1, 2): 29–48..
- [7]
^Linka M, Weber AP (2011). "Evolutionary Integration of Chloroplast Metabolism with the Metabolic Networks of the Cells". In Burnap RL, Vermaas WF. Functional Genomics and Evolution of Photosynthetic Systems. Springer. p. 215. ISBN 978-9400715332..
- [8]
^Marsh, Mark (2001). Endocytosis. Oxford University Press. p. vii. ISBN 978-0-19-963851-2..
- [9]
^Hetzer MW (March 2010). "The nuclear envelope". Cold Spring Harbor Perspectives in Biology. 2 (3): a000539. doi:10.1101/cshperspect.a000539. PMC 2829960. PMID 20300205..
- [10]
^"Endoplasmic Reticulum (Rough and Smooth)". British Society for Cell Biology. Retrieved 12 November 2017..
- [11]
^"Golgi Apparatus". British Society for Cell Biology. Retrieved 12 November 2017..
- [12]
^"Lysosome". British Society for Cell Biology. Retrieved 12 November 2017..
- [13]
^Mack, Steve (1 May 2006). "Re: Are there eukaryotic cells without mitochondria?". madsci.org..
- [14]
^Watson J, Hopkins N, Roberts J, Steitz JA, Weiner A (1988). "28: The Origins of Life". Molecular Biology of the Gene (Fourth ed.). Menlo Park, CA: The Benjamin/Cummings Publishing Company, Inc. p. 1154. ISBN 978-0-8053-9614-0..
- [15]
^Davis, Josh L. (13 May 2016). "Scientists Shocked To Discover Eukaryote With NO Mitochondria". IFL Science. Retrieved 2016-05-13..
- [16]
^Sato, N. (2006). "Origin and Evolution of Plastids: Genomic View on the Unification and Diversity of Plastids". In R.R. Wise; J.K. Hoober. The Structure and Function of Plastids. Advances in Photosynthesis and Respiration. 23. Springer Netherlands. pp. 75–102. doi:10.1007/978-1-4020-4061-0_4. ISBN 978-1-4020-4060-3..
- [17]
^Margulis L (1998). Symbiotic planet: a new look at evolution. New York: Basic Books. ISBN 978-0-465-07271-2. OCLC 39700477.[页码请求].
- [18]
^琳·马古利斯,希瑟·麦肯和洛林·奥伦德岑斯基(编辑。),图解协议词汇表琼斯和巴特利特出版社,波士顿,1993年,第十八页。ISBN 0-86720-081-2.
- [19]
^Vorobjev IA, Nadezhdina ES (1987). The centrosome and its role in the organization of microtubules. International Review of Cytology. 106. pp. 227–293. doi:10.1016/S0074-7696(08)61714-3. ISBN 978-0-12-364506-7. PMID 3294718..
- [20]
^Howland JL (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. pp. 69–71. ISBN 978-0-19-511183-5..
- [21]
^Fry, Stephen C. (1989). "The Structure and Functions of Xyloglucan". Journal of Experimental Botany. 40 (1): 1–11. doi:10.1093/jxb/40.1.1..
- [22]
^Raven J (July 1987). "The role of vacuoles". New Phytologist. 106 (3): 357–422. doi:10.1111/j.1469-8137.1987.tb00149.x..
- [23]
^Oparka K (2005). Plasmodesmata. Oxford, UK: Blackwell Publishing..
- [24]
^Raven PH, Evert RF, Eichorm SE (1999). Biology of Plants. New York: W.H. Freeman..
- [25]
^Silflow CD, Lefebvre PA (December 2001). "Assembly and motility of eukaryotic cilia and flagella. Lessons from Chlamydomonas reinhardtii". Plant Physiology. 127 (4): 1500–1507. doi:10.1104/pp.010807. PMC 1540183. PMID 11743094..
- [26]
^Deacon J (2005). Fungal Biology. Cambridge, Massachusetts: Blackwell Publishers. pp. 4 and passim. ISBN 978-1-4051-3066-0..
- [27]
^Keeling PJ (October 2004). "Diversity and evolutionary history of plastids and their hosts". American Journal of Botany. 91 (10): 1481–1493. doi:10.3732/ajb.91.10.1481. PMID 21652304..
- [28]
^David J. Patterson. "Amoebae: Protists Which Move and Feed Using Pseudopodia". Tree of Life Web Project. Retrieved 12 November 2017..
- [29]
^Lane N (June 2011). "Energetics and genetics across the prokaryote-eukaryote divide". Biology Direct. 6 (1): 35. doi:10.1186/1745-6150-6-35. PMC 3152533. PMID 21714941..
- [30]
^Dacks J, Roger AJ (June 1999). "The first sexual lineage and the relevance of facultative sex". Journal of Molecular Evolution. 48 (6): 779–783. Bibcode:1999JMolE..48..779D. doi:10.1007/PL00013156. PMID 10229582..
- [31]
^Ramesh MA, Malik SB, Logsdon JM (January 2005). "A phylogenomic inventory of meiotic genes; evidence for sex in Giardia and an early eukaryotic origin of meiosis". Current Biology. 15 (2): 185–191. doi:10.1016/j.cub.2005.01.003. PMID 15668177..
- [32]
^Malik SB, Pightling AW, Stefaniak LM, Schurko AM, Logsdon JM (August 2007). Hahn MW, ed. "An expanded inventory of conserved meiotic genes provides evidence for sex in Trichomonas vaginalis". PLOS One. 3 (8): e2879. Bibcode:2008PLoSO...3.2879M. doi:10.1371/journal.pone.0002879. PMC 2488364. PMID 18663385..
- [33]
^Akopyants NS, Kimblin N, Secundino N, Patrick R, Peters N, Lawyer P, Dobson DE, Beverley SM, Sacks DL (April 2009). "Demonstration of genetic exchange during cyclical development of Leishmania in the sand fly vector". Science. 324 (5924): 265–268. Bibcode:2009Sci...324..265A. doi:10.1126/science.1169464. PMC 2729066. PMID 19359589..
- [34]
^Lahr DJ, Parfrey LW, Mitchell EA, Katz LA, Lara E (July 2011). "The chastity of amoebae: re-evaluating evidence for sex in amoeboid organisms". Proceedings: Biological Sciences. 278 (1715): 2081–2090. doi:10.1098/rspb.2011.0289. PMC 3107637. PMID 21429931..
- [35]
^Moore RT (1980). "Taxonomic proposals for the classification of marine yeasts and other yeast-like fungi including the smuts". Botanica Marina. 23: 361–373..
- [36]
^Goldfuß (1818). "Ueber die Classification der Zoophyten" [On the classification of zoophytes]. Isis, oder, Encyclopädische Zeitung von Oken (in German). 2 (6): 1008–1019.CS1 maint: Unrecognized language (link)从第1008页开始:“厄斯特·克拉斯。荨麻。原生动物。”(头等舱。原始动物。原生动物。)[注:这本杂志每页的每一栏都有编号;每页有两列。].
- [37]
^Scamardella, JM (1999). "Not plants or animals: a brief history of the origin of Kingdoms Protozoa, Protista and Protoctista" (PDF). International Microbiology. 2: 207–221. Archived from the original (PDF) on 14 June 2011..
- [38]
^Rothschild LJ (1989). "Protozoa, Protista, Protoctista: what's in a name?". Journal of the History of Biology. 22 (2): 277–305. doi:10.1007/BF00139515. PMID 11542176..
- [39]
^Woese CR, Kandler O, Wheelis ML (June 1990). "Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya". Proceedings of the National Academy of Sciences of the United States of America. 87 (12): 4576–4579. Bibcode:1990PNAS...87.4576W. doi:10.1073/pnas.87.12.4576. PMC 54159. PMID 2112744..
- [40]
^Adl SM, Simpson AG, Farmer MA, Andersen RA, Anderson OR, Barta JR, et al. (2005). "The new higher level classification of eukaryotes with emphasis on the taxonomy of protists". The Journal of Eukaryotic Microbiology. 52 (5): 399–451. doi:10.1111/j.1550-7408.2005.00053.x. PMID 16248873..
- [41]
^Harper JT, Waanders E, Keeling PJ (January 2005). "On the monophyly of chromalveolates using a six-protein phylogeny of eukaryotes" (PDF). International Journal of Systematic and Evolutionary Microbiology. 55 (Pt 1): 487–496. doi:10.1099/ijs.0.63216-0. PMID 15653923. Archived from the original (PDF) on 17 December 2008..
- [42]
^Parfrey LW, Barbero E, Lasser E, Dunthorn M, Bhattacharya D, Patterson DJ, Katz LA (December 2006). "Evaluating support for the current classification of eukaryotic diversity". PLoS Genetics. 2 (12): e220. doi:10.1371/journal.pgen.0020220. PMC 1713255. PMID 17194223..
- [43]
^Adl SM, Simpson AG, Lane CE, Lukeš J, Bass D, Bowser SS, et al. (September 2012). "The revised classification of eukaryotes" (PDF). The Journal of Eukaryotic Microbiology. 59 (5): 429–93. doi:10.1111/j.1550-7408.2012.00644.x. PMC 3483872. PMID 23020233. Archived from the original (PDF) on 16 June 2016..
- [44]
^Burki F (May 2014). "The eukaryotic tree of life from a global phylogenomic perspective". Cold Spring Harbor Perspectives in Biology. 6 (5): a016147. doi:10.1101/cshperspect.a016147. PMC 3996474. PMID 24789819..
- [45]
^Zhao S, Burki F, Bråte J, Keeling PJ, Klaveness D, Shalchian-Tabrizi K (June 2012). "Collodictyon – an ancient lineage in the tree of eukaryotes". Molecular Biology and Evolution. 29 (6): 1557–1568. doi:10.1093/molbev/mss001. PMC 3351787. PMID 22319147..
- [46]
^Romari K, Vaulot D (2004). "Composition and temporal variability of picoeukaryote communities at a coastal site of the English Channel from 18S rDNA sequences". Limnol Oceanogr. 49 (3): 784–798. Bibcode:2004LimOc..49..784R. doi:10.4319/lo.2004.49.3.0784..
- [47]
^Roger AJ, Simpson AG (February 2009). "Evolution: revisiting the root of the eukaryote tree". Current Biology. 19 (4): R165–67. doi:10.1016/j.cub.2008.12.032. PMID 19243692..
- [48]
^Sanders, Robert (11 April 2016). "Wealth of unsuspected new microbes expands tree of life". Berkeley News. Retrieved 2016-04-11..
- [49]
^Zimmer C (11 April 2016). "Scientists Unveil New 'Tree of Life'". The New York Times. Retrieved 2016-04-11..
- [50]
^Tovar J, Fischer A, Clark CG (June 1999). "The mitosome, a novel organelle related to mitochondria in the amitochondrial parasite Entamoeba histolytica". Molecular Microbiology. 32 (5): 1013–1021. doi:10.1046/j.1365-2958.1999.01414.x. PMID 10361303..
- [51]
^Boxma B, de Graaf RM, van der Staay GW, van Alen TA, Ricard G, Gabaldón T, van Hoek AH, Moon-van der Staay SY, Koopman WJ, van Hellemond JJ, Tielens AG, Friedrich T, Veenhuis M, Huynen MA, Hackstein JH (March 2005). "An anaerobic mitochondrion that produces hydrogen" (PDF). Nature. 434 (7029): 74–79. Bibcode:2005Natur.434...74B. doi:10.1038/nature03343. PMID 15744302..
- [52]
^Burki F, Shalchian-Tabrizi K, Minge M, Skjaeveland A, Nikolaev SI, Jakobsen KS, Pawlowski J (August 2007). Butler G, ed. "Phylogenomics reshuffles the eukaryotic supergroups". PLOS One. 2 (8): e790. Bibcode:2007PLoSO...2..790B. doi:10.1371/journal.pone.0000790. PMC 1949142. PMID 17726520..
- [53]
^Burki F, Shalchian-Tabrizi K, Pawlowski J (August 2008). "Phylogenomics reveals a new 'megagroup' including most photosynthetic eukaryotes". Biology Letters. 4 (4): 366–369. doi:10.1098/rsbl.2008.0224. PMC 2610160. PMID 18522922..
- [54]
^Burki F, Inagaki Y, Bråte J, Archibald JM, Keeling PJ, Cavalier-Smith T, Sakaguchi M, Hashimoto T, Horak A, Kumar S, Klaveness D, Jakobsen KS, Pawlowski J, Shalchian-Tabrizi K (July 2009). "Large-scale phylogenomic analyses reveal that two enigmatic protist lineages, telonemia and centroheliozoa, are related to photosynthetic chromalveolates". Genome Biology and Evolution. 1: 231–238. doi:10.1093/gbe/evp022. PMC 2817417. PMID 20333193..
- [55]
^Hackett JD, Yoon HS, Li S, Reyes-Prieto A, Rümmele SE, Bhattacharya D (August 2007). "Phylogenomic analysis supports the monophyly of cryptophytes and haptophytes and the association of rhizaria with chromalveolates". Molecular Biology and Evolution. 24 (8): 1702–1713. doi:10.1093/molbev/msm089. PMID 17488740..
- [56]
^Jagus R, Bachvaroff TR, Joshi B, Place AR (2012). "Diversity of Eukaryotic Translational Initiation Factor eIF4E in Protists". Comparative and Functional Genomics. 2012: 1–21. doi:10.1155/2012/134839. PMC 3388326. PMID 22778692..
- [57]
^Burki F, Kaplan M, Tikhonenkov DV, Zlatogursky V, Minh BQ, Radaykina LV, Smirnov A, Mylnikov AP, Keeling PJ (January 2016). "Untangling the early diversification of eukaryotes: a phylogenomic study of the evolutionary origins of Centrohelida, Haptophyta and Cryptista". Proceedings: Biological Sciences. 283 (1823): 20152802. doi:10.1098/rspb.2015.2802. PMC 4795036. PMID 26817772..
- [58]
^Janouškovec J, Tikhonenkov DV, Burki F, Howe AT, Rohwer FL, Mylnikov AP, Keeling PJ (December 2017). "A New Lineage of Eukaryotes Illuminates Early Mitochondrial Genome Reduction". Current Biology. 27 (23): 3717–24.e5. doi:10.1016/j.cub.2017.10.051. PMID 29174886..
- [59]
^Bodył A (February 2018). "Did some red alga-derived plastids evolve via kleptoplastidy? A hypothesis". Biological Reviews of the Cambridge Philosophical Society. 93 (1): 201–222. doi:10.1111/brv.12340. PMID 28544184..
- [60]
^Brown MW, Heiss AA, Kamikawa R, Inagaki Y, Yabuki A, Tice AK, Shiratori T, Ishida KI, Hashimoto T, Simpson AG, Roger AJ (February 2018). "Phylogenomics Places Orphan Protistan Lineages in a Novel Eukaryotic Super-Group". Genome Biology and Evolution. 10 (2): 427–433. doi:10.1093/gbe/evy014. PMC 5793813. PMID 29360967..
- [61]
^Lax G, Eglit Y, Eme L, Bertrand EM, Roger AJ, Simpson AG (November 2018). "Hemimastigophora is a novel supra-kingdom-level lineage of eukaryotes". Nature. 564 (7736): 410–414. doi:10.1038/s41586-018-0708-8. PMID 30429611..
- [62]
^Baurain D, Brinkmann H, Petersen J, Rodríguez-Ezpeleta N, Stechmann A, Demoulin V, Roger AJ, Burger G, Lang BF & Philippe H (July 2010). "Phylogenomic evidence for separate acquisition of plastids in cryptophytes, haptophytes, and stramenopiles". Molecular Biology and Evolution. 27 (7): 1698–1709. doi:10.1093/molbev/msq059. PMID 20194427..
- [63]
^Burki F, Okamoto N, Pombert JF & Keeling PJ (June 2012). "The evolutionary history of haptophytes and cryptophytes: phylogenomic evidence for separate origins". Proceedings: Biological Sciences. 279 (1736): 2246–2254. doi:10.1098/rspb.2011.2301. PMC 3321700. PMID 22298847..
- [64]
^Cavalier-Smith T (2006). "Protist phylogeny and the high-level classification of Protozoa". European Journal of Protistology. 39 (4): 338–348. doi:10.1078/0932-4739-00002..
- [65]
^Burki F, Pawlowski J (October 2006). "Monophyly of Rhizaria and multigene phylogeny of unicellular bikonts". Molecular Biology and Evolution. 23 (10): 1922–1930. doi:10.1093/molbev/msl055. PMID 16829542..
- [66]
^Ren R, Sun Y, Zhao Y, Geiser D, Ma H, Zhou X (September 2016). "Phylogenetic Resolution of Deep Eukaryotic and Fungal Relationships Using Highly Conserved Low-Copy Nuclear Genes". Genome Biology and Evolution. 8 (9): 2683–2701. doi:10.1093/gbe/evw196. PMC 5631032. PMID 27604879..
- [67]
^Derelle R, Torruella G, Klimeš V, Brinkmann H, Kim E, Vlček Č, Lang BF, Eliáš M (February 2015). "Bacterial proteins pinpoint a single eukaryotic root". Proceedings of the National Academy of Sciences of the United States of America. 112 (7): E693–699. Bibcode:2015PNAS..112E.693D. doi:10.1073/pnas.1420657112. PMC 4343179. PMID 25646484..
- [68]
^Yang J, Harding T, Kamikawa R, Simpson AG, Roger AJ (May 2017). "Mitochondrial Genome Evolution and a Novel RNA Editing System in Deep-Branching Heteroloboseids". Genome Biology and Evolution. 9 (5): 1161–1174. doi:10.1093/gbe/evx086. PMC 5421314. PMID 28453770..
- [69]
^Cavalier-Smith T, Fiore-Donno AM, Chao E, Kudryavtsev A, Berney C, Snell EA, Lewis R (February 2015). "Multigene phylogeny resolves deep branching of Amoebozoa". Molecular Phylogenetics and Evolution. 83: 293–304. doi:10.1016/j.ympev.2014.08.011. PMID 25150787..
- [70]
^Torruella G, de Mendoza A, Grau-Bové X, Antó M, Chaplin MA, del Campo J, Eme L, Pérez-Cordón G, Whipps CM, Nichols KM, Paley R, Roger AJ, Sitjà-Bobadilla A, Donachie S, Ruiz-Trillo I (September 2015). "Phylogenomics Reveals Convergent Evolution of Lifestyles in Close Relatives of Animals and Fungi". Current Biology. 25 (18): 2404–2410. doi:10.1016/j.cub.2015.07.053. PMID 26365255..
- [71]
^López-García P, Eme L, Moreira D (December 2017). "Symbiosis in eukaryotic evolution". Journal of Theoretical Biology. 434: 20–33. doi:10.1016/j.jtbi.2017.02.031. PMC 5638015. PMID 28254477..
- [72]
^Ponce-Toledo RI, Deschamps P, López-García P, Zivanovic Y, Benzerara K, Moreira D (February 2017). "An Early-Branching Freshwater Cyanobacterium at the Origin of Plastids". Current Biology. 27 (3): 386–391. doi:10.1016/j.cub.2016.11.056. PMC 5650054. PMID 28132810..
- [73]
^de Vries J, Archibald JM (February 2017). "Endosymbiosis: Did Plastids Evolve from a Freshwater Cyanobacterium?". Current Biology. 27 (3): R103–105. doi:10.1016/j.cub.2016.12.006. PMID 28171752..
- [74]
^Cavalier-Smith T, Chao EE, Lewis R (April 2018). "Multigene phylogeny and cell evolution of chromist infrakingdom Rhizaria: contrasting cell organisation of sister phyla Cercozoa and Retaria". Protoplasma. 255 (5): 1517–1574. doi:10.1007/s00709-018-1241-1. PMC 6133090. PMID 29666938..
- [75]
^He D, Fiz-Palacios O, Fu CJ, Fehling J, Tsai CC, Baldauf SL (February 2014). "An alternative root for the eukaryote tree of life". Current Biology. 24 (4): 465–470. doi:10.1016/j.cub.2014.01.036. PMID 24508168..
- [76]
^Hug LA, Baker BJ, Anantharaman K, Brown CT, Probst AJ, Castelle CJ, Butterfield CN, Hernsdorf AW, Amano Y, Ise K, Suzuki Y, Dudek N, Relman DA, Finstad KM, Amundson R, Thomas BC, Banfield JF (April 2016). "A new view of the tree of life". Nature Microbiology. 1 (5): 16048. doi:10.1038/nmicrobiol.2016.48. PMID 27572647..
- [77]
^Retallack GJ, Krull ES, Thackray GD, Parkinson DH (2013). "Problematic urn-shaped fossils from a Paleoproterozoic (2.2 Ga) paleosol in South Africa". Precambrian Research. 235: 71–87. Bibcode:2013PreR..235...71R. doi:10.1016/j.precamres.2013.05.015..
- [78]
^El Albani A, Bengtson S, Canfield DE, Bekker A, Macchiarelli R, Mazurier A, Hammarlund EU, Boulvais P, Dupuy JJ, Fontaine C, Fürsich FT, Gauthier-Lafaye F, Janvier P, Javaux E, Ossa FO, Pierson-Wickmann AC, Riboulleau A, Sardini P, Vachard D, Whitehouse M, Meunier A (July 2010). "Large colonial organisms with coordinated growth in oxygenated environments 2.1 Gyr ago". Nature. 466 (7302): 100–104. Bibcode:2010Natur.466..100A. doi:10.1038/nature09166. PMID 20596019..
- [79]
^Bengtson S, Belivanova V, Rasmussen B, Whitehouse M (May 2009). "The controversial "Cambrian" fossils of the Vindhyan are real but more than a billion years older". Proceedings of the National Academy of Sciences of the United States of America. 106 (19): 7729–7734. Bibcode:2009PNAS..106.7729B. doi:10.1073/pnas.0812460106. PMC 2683128. PMID 19416859..
- [80]
^Brocks JJ, Logan GA, Buick R, Summons RE (August 1999). "Archean molecular fossils and the early rise of eukaryotes". Science. 285 (5430): 1033–1036. CiteSeerX 10.1.1.516.9123. doi:10.1126/science.285.5430.1033. PMID 10446042..
- [81]
^Ward P (9 Feb 2008). "Mass extinctions: the microbes strike back". New Scientist: 40–43..
- [82]
^French KL, Hallmann C, Hope JM, Schoon PL, Zumberge JA, Hoshino Y, Peters CA, George SC, Love GD, Brocks JJ, Buick R, Summons RE (May 2015). "Reappraisal of hydrocarbon biomarkers in Archean rocks". Proceedings of the National Academy of Sciences of the United States of America. 112 (19): 5915–5920. Bibcode:2015PNAS..112.5915F. doi:10.1073/pnas.1419563112. PMC 4434754. PMID 25918387..
- [83]
^Isson TT, Love GD, Dupont CL, Reinhard CT, Zumberge AJ, Asael D, et al. (June 2018). "Tracking the rise of eukaryotes to ecological dominance with zinc isotopes". Geobiology. 16 (4): 341–352. doi:10.1111/gbi.12289. PMID 29869832..
- [84]
^Yoshikawa G, Blanc-Mathieu R, Song C, Kayama Y, Mochizuki T, Murata K, Ogata H, Takemura M (April 2019). "Medusavirus, a Novel Large DNA Virus Discovered from Hot Spring Water". Journal of Virology. 93 (8). doi:10.1128/JVI.02130-18. PMC 6450098. PMID 30728258. Lay summary – EurekAlert! (30 April 2019)..
- [85]
^Martin W (December 2005). "Archaebacteria (Archaea) and the origin of the eukaryotic nucleus". Current Opinion in Microbiology. 8 (6): 630–637. doi:10.1016/j.mib.2005.10.004. PMID 16242992..
- [86]
^Bell PJ (September 2001). "Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus?". Journal of Molecular Evolution. 53 (3): 251–256. Bibcode:2001JMolE..53..251L. doi:10.1007/s002390010215. PMID 11523012..
- [87]
^Takemura M (May 2001). "Poxviruses and the origin of the eukaryotic nucleus". Journal of Molecular Evolution. 52 (5): 419–425. Bibcode:2001JMolE..52..419T. doi:10.1007/s002390010171. PMID 11443345..
- [88]
^Wächtershäuser G (January 2003). "From pre-cells to Eukarya – a tale of two lipids". Molecular Microbiology. 47 (1): 13–22. doi:10.1046/j.1365-2958.2003.03267.x. PMID 12492850..
- [89]
^Wächtershäuser G (October 2006). "From volcanic origins of chemoautotrophic life to Bacteria, Archaea and Eukarya". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 361 (1474): 1787–1806, discussion 1806–1808. doi:10.1098/rstb.2006.1904. PMC 1664677. PMID 17008219..
- [90]
^Lane, Nick (2016). The Vital Question: Why is Life the Way it is? (paperback ed.). Profile Books. pp. 157–91. ISBN 978-1-781-25037-2..
- [91]
^Hartman H, Fedorov A (February 2002). "The origin of the eukaryotic cell: a genomic investigation". Proceedings of the National Academy of Sciences of the United States of America. 99 (3): 1420–1425. Bibcode:2002PNAS...99.1420H. doi:10.1073/pnas.032658599. PMC 122206. PMID 11805300..
- [92]
^Harish A, Tunlid A, Kurland CG (August 2013). "Rooted phylogeny of the three superkingdoms". Biochimie. 95 (8): 1593–1604. doi:10.1016/j.biochi.2013.04.016. PMID 23669449..
- [93]
^Harish A, Kurland CG (July 2017). "Akaryotes and Eukaryotes are independent descendants of a universal common ancestor". Biochimie. 138: 168–183. doi:10.1016/j.biochi.2017.04.013. PMID 28461155..
- [94]
^Spang A, Saw JH, Jørgensen SL, Zaremba-Niedzwiedzka K, Martijn J, Lind AE, van Eijk R, Schleper C, Guy L, Ettema TJ (May 2015). "Complex archaea that bridge the gap between prokaryotes and eukaryotes". Nature. 521 (7551): 173–179. Bibcode:2015Natur.521..173S. doi:10.1038/nature14447. PMC 4444528. PMID 25945739..
- [95]
^Harish A, Kurland CG (July 2017). "Empirical genome evolution models root the tree of life". Biochimie. 138: 137–155. doi:10.1016/j.biochi.2017.04.014. PMID 28478110..
- [96]
^Jékely G (2007). "Origin of Eukaryotic Endomembranes: A Critical Evaluation of Different Model Scenarios". Eukaryotic Membranes and Cytoskeleton. Advances in Experimental Medicine and Biology. 607. pp. 38–51. doi:10.1007/978-0-387-74021-8_3. ISBN 978-0-387-74020-1. PMID 17977457..
- [97]
^Cavalier-Smith T (March 2002). "The phagotrophic origin of eukaryotes and phylogenetic classification of Protozoa". International Journal of Systematic and Evolutionary Microbiology. 52 (Pt 2): 297–354. doi:10.1099/00207713-52-2-297. PMID 11931142..
- [98]
^Martin W, Müller M (March 1998). "The hydrogen hypothesis for the first eukaryote". Nature. 392 (6671): 37–41. Bibcode:1998Natur.392...37M. doi:10.1038/32096. PMID 9510246..
- [99]
^Pisani D, Cotton JA, McInerney JO (August 2007). "Supertrees disentangle the chimerical origin of eukaryotic genomes". Molecular Biology and Evolution. 24 (8): 1752–1760. doi:10.1093/molbev/msm095. PMID 17504772..
- [100]
^Latorre A, Durban A, Moya A, Pereto J (2011). "The role of symbiosis in eukaryotic evolution". In Gargaud M, López-Garcìa P, Martin H. Origins and Evolution of Life: An Astrobiological Perspective. Cambridge: Cambridge University Press. pp. 326–339. ISBN 978-0-521-76131-4..
- [101]
^Ayala J (April 1994). "Transport and internal organization of membranes: vesicles, membrane networks and GTP-binding proteins". Journal of Cell Science. 107 ( Pt 4) (107): 753–763. PMID 8056835..
- [102]
^Martin WF. "The Origin of Mitochondria". Scitable. Nature education. Retrieved 2013-03-27..
- [103]
^Dacks JB, Field MC (August 2018). "Evolutionary origins and specialisation of membrane transport". Current Opinion in Cell Biology. 53: 70–76. doi:10.1016/j.ceb.2018.06.001. PMC 6141808. PMID 29929066..
- [104]
^Martijn J, Vosseberg J, Guy L, Offre P, Ettema TJ (May 2018). "Deep mitochondrial origin outside the sampled alphaproteobacteria". Nature. 557 (7703): 101–105. doi:10.1038/s41586-018-0059-5. PMID 29695865. Lay summary – The Scientist..
- [105]
^Baum DA, Baum B (October 2014). "An inside-out origin for the eukaryotic cell". BMC Biology. 12: 76. doi:10.1186/s12915-014-0076-2. PMC 4210606. PMID 25350791. Lay summary – University of Wisconsin-Madison..
- [106]
^Brouwers L (12 April 2013). "How genetic plunder transformed a microbe into a pink, salt-loving scavenger". Scientific American..
- [107]
^Margulis L (1970). Origin of Eukaryotic Cells. New Haven, London: Yale University Press..
- [108]
^Margulis L (1993). Symbiosis in Cell Evolution. New York: W.H. Freeman..
- [109]
^Margulis L, Dolan MF, Guerrero R (June 2000). "The chimeric eukaryote: origin of the nucleus from the karyomastigont in amitochondriate protists". Proceedings of the National Academy of Sciences of the United States of America. 97 (13): 6954–6959. Bibcode:2000PNAS...97.6954M. doi:10.1073/pnas.97.13.6954. PMC 34369. PMID 10860956..
- [110]
^Moreira D, Lopez-Garcia P (November 1998). "Symbiosis between methanogenic archaea and delta-proteobacteria as the origin of eukaryotes: the syntrophic hypothesis". Journal of Molecular Evolution. 47 (5): 517–530. Bibcode:1998JMolE..47..517M. doi:10.1007/PL00006408. PMID 9797402..
- [111]
^López-García P, Moreira D (May 2006). "Selective forces for the origin of the eukaryotic nucleus". BioEssays. 28 (5): 525–533. doi:10.1002/bies.20413. PMID 16615090..
- [112]
^研究人员重新思考复杂细胞的祖先——量子杂志.
- [113]
^Pittis AA, Gabaldón T (March 2016). "Late acquisition of mitochondria by a host with chimaeric prokaryotic ancestry". Nature. 531 (7592): 101–104. Bibcode:2016Natur.531..101P. doi:10.1038/nature16941. PMC 4780264. PMID 26840490..
- [114]
^Burton ZF (1 August 2017). Evolution since Coding: Cradles, Halos, Barrels, and Wings. Academic Press. ISBN 9780128130346..
- [115]
^Martin WF, Roettger M, Ku C, Garg SG, Nelson-Sathi S, Landan G (February 2017). "Late Mitochondrial Origin Is an Artifact". Genome Biology and Evolution. 9 (2): 373–379. doi:10.1093/gbe/evx027. PMC 5516564. PMID 28199635..
暂无