物种
编辑生物学包含八大分类等级,一个域包含一个或多个界。中间次要的分类未显示。
在生物学中,物种是有机体的基本分类单位和分类等级,也是生物多样性的单位。物种通常被定义为最大的有机体群体,其中任何两个适当性别或交配类型的个体都能产生可育后代,这通常是通过有性繁殖来实现。定义物种的其他方法包括它们的核型、DNA序列、形态、行为或生态位。此外,古生物学家使用时序种的概念,因为化石不能用于检验繁殖。虽然这些定义似乎足够了,但如果仔细观察,它们代表的物种概念还存在问题。例如,在数百个相似微物种的物种复合体和环状物种中,亲缘关系紧密的物种之间的边界随着杂交变得不明确。此外,在只有无性繁殖的生物中,繁殖物种的概念被打破,每个克隆都可能是一个小种。
所有物种(除了病毒)都有一个由两部分组成的名字,即“双名”。双名的第一部分是该物种所属的属。第二部分被称为种名或种加词(用于植物学术语中,有时也用于动物学术语中)。例如,蟒蛇是蟒蛇属的四种动物之一。
这些都不是完全令人满意的定义,尽管理论上具有困难,但是科学家和保护主义者需要一个物种定义,让他们能够工作。如果物种是固定不变的,并且彼此明显不同,那就没有问题了,但是进化过程会导致物种不断地变化,相互之间也会变化。
从亚里士多德时代到18世纪,物种被视为固定的种类,可以被安排在一个等级体系中,即存在之链中。在19世纪,生物学家认识到,只要有足够的时间,物种就会进化。查尔斯·达尔文1859年的著作《物种起源》解释了物种是如何通过自然选择产生的。这种理解在20世纪通过遗传学和种群生态学得到了极大的扩展。遗传变异源于突变和重组,而生物体本身是可移动的,在不同的选择压力下导致地理隔离和遗传漂变。基因有时可以通过水平基因转移在物种之间交换;新物种可以通过杂交和多倍体迅速出现;物种可能会因为各种原因灭绝。病毒是一种特殊的情况,由突变和选择的平衡驱动,可以被视为准物种。
目录编辑
1 定义编辑
生物学家和分类学家已经做了许多定义物种的尝试,从形态学开始,向遗传学发展。像林奈这样的早期分类学家别无选择,只能描述他们所看到的:这后来被正式确定为类型学或形态学物种概念。恩斯特·迈尔强调生殖隔离,但这和其他物种概念一样,很难甚至不可能验证。[1][2] 后来的生物学家试图用认知和凝聚力等概念来完善迈尔的定义。[3] 许多概念非常相似或重叠,因此不容易计数:生物学家R. L. 梅登记录了大约24个概念,[4] 科学哲学家约翰·威尔金斯记录了26个概念。[1]威尔金斯进一步将物种概念分为七种基本概念:(1)基于无性繁殖生物的无性种(2)基于生物生殖隔离的生物学物种(3)基于生态位的生态学物种(4)基于谱系的进化学物种(5)基于基因库的遗传学物种(6)基于形式或表型的形态学物种(7)分类学物种,即由分类学家确定的物种[5]
1.1 类型学或形态学物种
类型物种是一组生物,其中个体符合某些固定的属性(类型),因此即使是没有受过教育的人也经常能像现代分类学家一样识别出相同的分类单元。[6][7] 标本中的变异或表型簇(如较长或较短的尾巴)可以区分物种。这种方法被用作确定物种的“经典”方法,例如在进化论早期的林奈。然而,不同的表型不一定是不同的物种(例如,双翼母亲所生的四翼果蝇不是不同的物种)。以这种方式命名的物种称为形态学物种。[8][9]
20世纪70年代,罗伯特·索卡尔(Robert R. Sokal)、西奥多·克罗韦洛(Theodore J. Crovello)和彼得·斯奈斯(Peter Sneath)对此提出了一种变体,一种表现型物种,定义为一组表型彼此相似,但与其他组的生物表型不同的生物。[10] 它与形态物种概念的不同之处在于,它包含了基于大量表型性状的多元比较的聚类实体的距离或相似性的数值度量。[11]
1.2 识别和内聚物种
配偶识别物种是一组有性繁殖的有机体,它们将彼此识别为潜在的配偶。[12][13]在此基础上扩展到允许交配后隔离后,内聚力物种成为最具包容性的群体,通过内在内聚的机制具有表型内聚的潜力;无论种群能否成功杂交,如果杂交的数量不足以完全混合它们各自的基因库。[14]它们仍然是不同的内聚物种。物种的生物概念提供了物种识别概念的进一步发展。[15]
1.3 遗传相似性和条形码物种
在微生物学中,基因甚至可以在远亲细菌之间自由移动,甚至可能扩展到整个细菌领域。根据经验,微生物学家认为,具有16S核糖体RNA基因序列相似度超过97%的细菌或古细菌,需要通过DNA-DNA杂交检验它们是否属于同一物种。[16]这一概念在2006年缩小到98.7%。[17]
DNA-DNA杂交已经过时,结果有时会导致对物种的误导性结论,就像中贼鸥和大贼鸥一样。[18][19] 现代的方法是使用计算来比较序列相似性。[20]
DNA条形码已经作为一种区分物种的方法被提出,甚至适合非专业人员使用。[21]所谓的条形码是细胞色素c氧化酶基因中线粒体基因的一个区域。一个名为“生命条形码数据系统(BOLD)”的数据库包含了来自190,000多个物种的DNA条形码序列。[22][23] 然而,像罗布·德萨勒这样的科学家已经表达了他们的担忧,他们认为传统分类学和DNA条形码(他们认为这是用词不当)需要调和,因为它们对物种的界定不同。[24] 由内共生体和其他载体介导的遗传渗入会进一步使条形码在物种鉴定中无效。[25]
1.4 系统发育、分支或进化学物种
系统发育或分支物种是进化上有分歧的谱系,它在时间和空间上保持了遗传完整性。[26][27][28] 分支物种是可以通过一组独特的形态或遗传特征来区分的最小群体。分子标记可用于确定不同物种细胞核或线粒体基因的遗传相似性。[27][29][30] 例如,在一项关于真菌的研究中,在所有研究概念中,使用分支物种研究核苷酸特征,在识别众多真菌物种方面产生了最准确的结果。[30][31] 系统发育物种概念的不同版本可能强调单系或可诊断性。[32]
与生物物种概念不同,分支物种不依赖生殖隔离,因此它独立于其他概念中这一不可分割的过程。[29] 它适用于无性谱系,并能检测最近的分化,而形态学物种概念却不能。[27][30] 然而,它并不是在所有情况下都有效,可能需要一个以上的多态位点才能给出准确的结果。[30] 这一概念可能导致现有物种,例如牛科(Bovidae),分裂出许多新的物种。[33][34][35]
乔治·盖洛德·辛普森在1951年提出的进化物种是“一个由有机体组成的实体,它通过时间和空间与其他此类实体保持其同一性,并有自己独立的进化命运和历史趋势”。[4][36] 这不同于生物物种的概念,体现了随时间推移的持久性。威利和梅登表示,他们认为进化物种概念与威利·亨宁(Willi Hennig)的物种血统概念“相同”,并断言生物物种概念、系统发育物种概念的“几个版本”,以及物种与高等分类群属于同一种类的想法不适合生物多样性研究(目的是准确估计物种数量)。他们进一步指出,这个概念对无性繁殖和有性繁殖物种都有效。[37]
1.5 生态学物种
生态物种是适应环境中某一特定资源(称为生态位)的一组有机体。根据这个概念,种群形成了离散的物候群,我们将其视为物种,因为控制资源分配的生态和进化过程往往会产生这些物候群。[38]
1.6 遗传学物种
罗伯特·贝克和罗伯特·布拉德利定义的遗传学物种是一组遗传隔离的杂交种群。这类似于迈尔的生物物种概念,但强调遗传隔离而不是生殖隔离。在21世纪,可以通过比较DNA序列来建立一个遗传物种,但是其他的方法在更早的时候就有了,例如比较核型(染色体组)和等位酶(酶变体)。[39]
1.7 进化上得重要单位
进化意义上的重要单位(ESU)或“野生动物物种”[40] 是为了保护目的而被认为是独特的生物体种群。[41]
1.8 时序钟
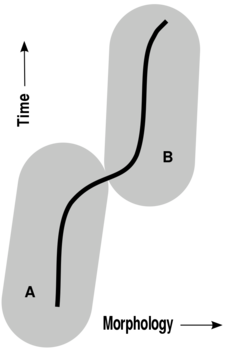
在古生物学中,只有化石的比较解剖学(形态学)作为证据,才能应用时序种的概念。在进化过程中(进化,不一定涉及分支),古生物学家试图确定一支物种序列,每一个物种都通过连续、缓慢和或多或少一致的变化,从之前已经灭绝的物种中衍生而来。在这样一个时间序列中,古生物学家评估一种形态上不同的物种需要多少变化才能被认为是与其祖先不同的物种。[42][43][44][45]
1.9 病毒准物种
病毒拥有庞大的种群,由于它们仅由蛋白质外壳中的一串DNA或RNA组成,所以很难认为它们是活的,并且变异迅速。所有这些因素使得传统的物种概念在很大程度上不适用。[46] 病毒准种是相似突变相关的一组基因型,在高度诱变的环境中竞争,因此受突变选择平衡的控制。据预测,在适应度景观中处于较低但进化中性和高度连接(即平坦)区域的病毒准物种将胜过位于较高但较窄适应度峰值的准物种,在该峰值中周围的突变体是不适合的,“准物种效应”或“平者生存”。没有迹象表明病毒准物种与传统的生物物种相似。[47][48][49]
2 分类和命名编辑
2.1 俗名和学名
各种生物的俗名往往含糊不清:“猫”可以指家猫(Felis catus),也可以指猫科动物(Felidae)。通用名称的另一个问题是它们经常因地而异,因此美洲豹、美洲狮、山猫、豹、油漆工和山狮在美国各地都表示美洲狮(Puma concolor),而“panther”也可能表示拉丁美洲的美洲虎(Panthera onca)或非洲和亚洲的花豹(Panthera pardus)。相比之下,物种的学名在被选定时是独一无二和通用的;它们同时由两部分组成:属名Puma和种加词concolor。[50][51]
2.2 物种描述
当模式标本被正式描述时,一个物种就被赋予了一个分类学名称,并在一份出版物中被赋予一个独特的学名。该描述通常提供识别新物种的手段,将其与其他先前描述的和相关的或易混淆的物种区分开来,并在论文被接受出版时提供有效发表的名称(植物学)或可用名(动物学)。这种类型的材料通常保存在一个永久的仓库里,通常是一个主要博物馆或大学的研究收藏,这使得独立的验证和比较标本的方法成为可能。[52][53][54]新物种的描述者被要求选择用《国际动物命名法》的话来说“合适、紧凑、悦耳、难忘且不会引起冒犯”的名称。[55]
2.3 缩写
书籍和文章有时故意不完全标明物种并使用缩写“sp.”单数或“spp.”(代表species pluralis,拉丁语代表物种的复数)用复数代替特定的名称或修饰语(例如Canis sp)。这通常发生在作者确信某些个体属于某个特定的属,但不确定他们属于哪个确切的物种时,这在古生物学中很常见。作者也可以使用“spp.”作为一种简短的说法,代表某事物适用于一个属内的许多物种,但并不适用于所有物种。如果科学家的意思是某事物适用于一个属内的所有物种,他们会使用属名而不用具体的名称或绰号。属和种的名称通常用斜体印刷。缩写,如“sp.”不应该大写。当一个物种的身份不清楚时,专家可以在这个称谓前使用“cf.”来表示需要确认。缩写“nr.”(near)或“aff.”(affine)可以在身份不清楚但物种看起来与后面提到的物种相似时使用。[56]
2.4 识别码
随着在线数据库的兴起,已经设计了编码来为已经定义的物种提供标识符,包括:
- 国家生物技术信息中心(NCBI)使用的是一个数字的“taxid”或者称为分类标识符,一个“稳定的唯一标识符”,例如智人的taxid是9606。[57]
- 京都基因和基因组百科全书(KEGG)对有限数量的生物体采用三个或四个字母的代码;例如,在这段代码中,智人就是简单的hsa。[58]
- UniProt使用不超过五个字母数字字符的“有机体助记符”,例如HUMAN代表智人。[59]
- 综合分类信息系统(ITIS)为每个物种提供了一个独特的数字。智人的LSID是urn:lsid:catalogueoflife.org:taxon:4da6736d-d35f-11e6-9d3f-bc764e092680:col20170225。[60]
2.5 统合和分割
一个特定物种的命名,包括它属于哪一个属(和更高的分类群),是关于该组生物体的进化关系和可区分性的假设。随着更多信息的到来,这一假设可能会得到证实或反驳。有时,尤其是在过去交流更加困难的时候,独立工作的分类学家给后来被确定为同一物种的个体生物起了两个不同的名字。当两个命名的物种被发现属于同一物种时,旧的物种名称被优先考虑并通常被保留,而新的名称被认为是低级同义词,这一过程被称为整合。将一个分类单元分成多个分类单元(通常是新的)称为分裂。分类学家通常被他们的同事称为“统合者”或“分割者”,这取决于他们识别有机体之间差异或共性的个人方法。[61][62][56]
2.6 广义和狭义
指导物种命名的命名规范,包括动物命名的ICZN和植物命名的ICN,并没有制定定义物种边界的规则。基于新的证据,研究可以改变界限,或者称为定义。物种可能需要用已有的边界定义来区分,在这种情况下,名称可以用狭义来限定,以表示作者(如命名物种的人)给出的确切含义,而反义词广义表示更广泛的用法,例如包括其他亚种。其他缩写,如“auct.”。(作者)和限定词如“non”(非)可用于进一步阐明特定作者描绘或描述物种的意义。[56][63][64]
3 迈尔的生物物种概念编辑
4 物种难题编辑
很难用适用于所有生物的方式来定义一个物种。[71] 关于物种划分的争论被称为物种问题。[72][72][73][74] 甚至在1859年,达尔文在《物种起源》中写道: 没有一个定义能让所有博物学家满意;然而,每一位博物学家在谈到一个物种时,都模糊地知道自己的意思。一般来说,这个术语包括一个独特的创造行为的未知元素。
4.1 当迈尔的概念崩溃时
按照迈尔的概念,一个简单的教科书定义对大多数多细胞生物都适用,但在几种情况下会崩溃:
- 当生物体无性繁殖时,如在单细胞生物体如细菌和其他原核生物中,[75] 以及单性生殖或无融合生殖的多细胞生物体中。[76][77][78]准物种一词有时用于快速突变实体如病毒等。[79][80]
- 当科学家不知道两个形态相似的有机体群体是否能够杂交时;古生物学中所有灭绝的生物都是这种情况,因为繁殖实验是不可能的。[81]
- 当杂交允许物种间大量基因流动时。[82]
- 在环形种中,当在广泛连续分布范围内的相邻种群成员成功杂交,但更远种群成员不成功杂交。[83]
柳莺和赤霞珠外形几乎相同,但不杂交。
分子和形态学研究之间的不一致使得物种鉴定变得困难;这些可以分为两种类型:(一)一种形态,多种谱系(例如形态趋同,隐种)和(二)一种谱系,多种形态(例如表型可塑性,多生命周期阶段)。[84] 此外,水平基因转移(HGT)使得很难定义一个物种。 所有物种的定义都假设一个生物体从一个或两个非常像“女儿”的父母那里获得基因,但这并不是HGT的情况。[85] 有强有力的证据表明,HGT存在于非常不同的原核生物群体之间,至少偶尔存在于不同的真核生物群体之间, 包括一些甲壳类动物和棘皮动物.[86]
进化生物学家詹姆斯·马利特得出结论:
没有简单的方法来区分相关的地理或时间形式是属于同一物种还是不同物种。物种差距只能在当地和某个时间点得到证实。人们不得不承认达尔文的观点是正确的:在很大的地理范围和时间段内,任何当地的现实或物种的完整性都会大大降低。
4.2 小种的聚集
物种概念因小种、包括许多植物在内的生物群体的存在而进一步削弱,这些生物群体遗传变异很小,通常形成物种复合体。[87]例如,蒲公英和黑莓是由许多小种聚集而成的——黑莓可能有400种,蒲公英有200多种,[88] 杂交、无融合生殖和多倍体使种群间的基因流难以确定,它们的分类学也存在争议。[89][90][91] 物种复合物存在于昆虫中,如袖蝶属的蝴蝶,[92]脊椎动物如树蛙属的树蛙,[93] 真菌,如蛤蟆菌。[94]
黑莓属于数以百计的灌木树莓种类集合体中的任何一种。
螺旋蝴蝶属包含许多相似的种类。
海泡石-条石斑鱼物种复合体包含至少六种树蛙。
4.3 杂交
自然杂交对生殖隔离物种的概念提出了挑战,因为可育杂种允许两个群体之间的基因流动。例如,小嘴乌鸦Corvus corone和冠小嘴乌鸦Corvus cornix出现并被分类为不同的物种,但它们在地理范围重叠的地方自由杂交。[95]
- 腐肉和带帽乌鸦的杂交允许基因在“物种”之间流动。
腐肉乌鸦
深色腹部、深灰色颈背的杂交种类
黑肚皮混血儿
蒙面乌鸦
4.4 环形种
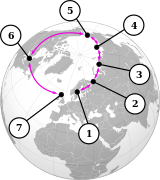
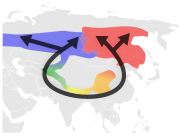
环形种是一系列相连的相邻种群,每个种群都可以与相邻的相关种群进行杂交,但在这个系列中至少存在两个“末端”种群,它们与杂交的关系过于遥远,尽管每个“相连”种群之间存在潜在的基因流动。[96] 这种非繁殖的但遗传上有联系的“末端”种群可能在同一个区域共存,从而关闭了环。因此,环形种对于任何依赖生殖隔离的物种概念来说都是一个难题。[97] 然而,环形种充其量是罕见的。提出的例子包括北极附近的银鸥-小黑背鸥复合体、美国的19种蝾螈组成的埃氏剑螈群,[98] 和亚洲的暗绿柳莺,[99] 但许多所谓的环形种被证明是错误分类的结果,导致人们质疑是否真的有环形种。[100][101][102][103]
七种海鸥在北极圈内繁殖。
环的两端:挪威的一只银鸥(银鸥)(前面)和一只较小的黑背鸥(银鸥)。
一种绿色的莺,圆齿黄莺。
喜马拉雅山周围五种绿莺的推测进化
5 变化编辑
无论是进化成新物种,[104] 与其他物种交换基因,[105] 与其他物种融合或者灭绝[106]物种都会发生变化。
5.1 物种形成
作为物种,生物种群进化成独特的或繁殖上孤立的进化过程称为物种形成。[107][108] 查尔斯·达尔文在他1859年的著作《物种起源》中首次描述了自然选择在物种形成中的作用。[109] 物种形成依赖于生殖隔离的措施,即基因流的减少。这在异源物种形成中最容易发生,在异源物种形成中,种群在地理上是分开的,并且随着突变的积累而逐渐分化。生殖隔离受到杂交的威胁,但正如巴特森-多勃赞斯基-穆勒模型[104] 所描述的,一旦一对群体具有相同基因的不相容等位基因,就可以选择这种隔离。一种不同的机制,即系统物种形成,是指一个谱系随着时间的推移逐渐转变成一种新的和独特的形式,而不增加最终物种的数量。[110]
5.2 物种间的基因交换
不同物种的生物体之间通过杂交、抗原转移或重组进行的水平基因转移,有时是遗传变异的重要来源。病毒可以在物种之间转移基因。细菌可以与其他物种的细菌交换质粒,包括不同系统发育领域中一些明显远亲的细菌,这使得对它们之间关系的分析变得困难,并削弱了细菌物种的概念。[111][112][112][105]
路易斯-玛丽·博比和霍华德·奥奇曼根据对许多细菌基因组的分析,认为它们通常可以被归类为“定期交换基因的群落”,就像植物和动物可以被归类为繁殖隔离的繁殖种群一样。细菌因此可以形成物种,类似于迈尔的生物物种概念,由通过同源重组交换基因的无性繁殖群体组成。[113][114]
5.3 灭绝
当一个物种的最后一个个体死亡时,这个物种就灭绝了,但是它可能在那个时刻之前就已经在功能上灭绝了。据估计,地球上有超过99%的物种,大约50亿种,现在已经灭绝。其中一些处于大规模灭绝中,如二叠纪、三叠纪和白垩纪末期。大规模灭绝有多种原因,包括火山活动、气候变化以及海洋和大气化学的变化,它们反过来又对地球的生态、大气、地表和水域产生重大影响。[115][116] 另一种形式的灭绝是通过杂交一个物种被另一个物种同化。由此产生的单一物种被称为“复合物种”。[117]
6 实际影响编辑
生物学家和自然资源保护主义者需要在工作过程中对生物体进行分类和识别。将生物可靠地分配给一个物种的困难对研究结果的有效性构成了威胁,例如,对一个物种在生态系统中的丰富度的测量没有实际意义。使用系统发育物种概念的调查报告了比那些使用非系统发育概念的物种多48%的物种,相应的种群和范围更小;这被称为“分类学膨胀”,[118]这可能导致濒危物种数量的虚假变化,并随之带来政治和实际困难。[119][120] 一些观察家声称,理解物种形成过程的愿望与识别和分类的需要之间存在内在冲突。[120]
许多国家的保护法律都有防止物种灭绝的特殊规定。两个物种之间的杂交带,一个受保护,一个不受保护,有时会导致立法者、土地所有者和自然资源保护者之间的冲突。北美的一个典型案例是受保护的北方斑点猫头鹰,它与未受保护的加利福尼亚斑点猫头鹰和横斑猫头鹰杂交;这导致了法律辩论。[121] 有人认为,物种问题是由物种概念的各种用途造成的,解决办法是放弃它和所有其他分类等级,而是使用无等级的单系群。也有人认为,由于物种是不可比较的,对它们进行计数并不是生物多样性的有效衡量标准;已经提出了系统发育生物多样性的替代措施。[122][123]
7 历史编辑
7.1 古典形式
在他的生物学中,亚里斯多德用γένος (génos)一词来表示一种动物,如鸟或鱼,而εἶδος (eidos)则表示一种动物中的特定形式,如(鸟类中的)鹤、鹰、乌鸦或麻雀。这些术语被翻译成拉丁语中的“属”和“种”,尽管它们与林奈的术语不一致。今天,鸟是一个纲,鹤是一个科,乌鸦是一个属。一种因其属性而不同;例如,一只鸟有羽毛、喙、翅膀、硬壳蛋和温暖的血液。一种形式的区别在于它的所有成员共享,后代从父母那里继承了他们可能有的任何变异。亚里士多德认为所有的种类和形式都是独特而不变的。他的方法在文艺复兴之前一直很有影响力。[124]
7.2 固定物种
当现代早期的观察者开始发展生物的组织系统时,他们把每种动物或植物放在一个环境中。许多早期的描绘方案现在被认为是异想天开:方案包括基于颜色(所有黄花植物)或行为(蛇、蝎子和某些咬人的蚂蚁)的血缘关系。英国博物学家约翰·雷于1686年首次尝试对物种进行生物学定义,具体如下:
在我看来,确定物种的最可靠标准莫过于从种子繁殖中延续自身的显著特征。因此,无论个体或物种发生什么变化,如果它们来自同一种植物的种子,它们都是偶然的变化,不能用来区分一个物种...同样不同的动物会永久保持它们独特的物种;一个物种永远不会从另一个物种的种子中诞生,反之亦然。
在我看来,确定物种的最可靠标准莫过于从种子繁殖中延续自身的显著特征。因此,无论在个体或物种中发生什么样的变异,如果它们来自同一种植物的种子,它们都是偶然的变异,而不是为了区分一个物种...同样,不同的动物会永久地保存它们独特的物种;一个物种永远不会从另一个物种的种子中产生,反之亦然。[125]
在18世纪,瑞典科学家卡尔·林奈(Carl Linnaeus)根据共同的身体特征对生物体进行分类,而不仅仅是基于差异。[126] 他基于可观察到的特征建立了分类层次的概念,旨在反映自然关系。[127][128] 然而,当时人们仍然普遍认为物种之间没有有机联系,不管它们看起来多么相似。这种观点受到欧洲学术和宗教教育的影响,认为生命的类别是由上帝决定的,形成了亚里士多德的等级制度,即自然等级或伟大的存在链。然而,不管它是否应该是固定的,梯阶(梯子)内在地暗示着攀爬的可能性。[129]
7.3 可变性
在观察杂交的证据时,林奈认识到物种不是固定的,可能会改变;他不认为新物种会出现,也不认为神圣的固定物种会通过杂交或驯化过程而改变。[130] 到了19世纪,博物学家认识到物种会随着时间的推移而改变形态,地球的历史为重大变化提供了足够的时间。让-巴蒂斯特·拉马克在他1809年的《动物哲学》中描述了物种的嬗变,提出物种可以随着时间的推移而改变,这与亚里士多德的思想截然不同。[131]
1859年,查尔斯·达尔文和阿尔弗雷德·罗素·华莱士对进化和新物种的形成做了令人信服的描述。达尔文认为是种群,而不是个体,通过自然选择从个体间自然发生的变异进化而来。[132] 这需要一个新的物种定义。达尔文得出结论,物种就像它们看起来的样子:对于暂时用于命名相互作用的个体的群体的想法,写道:
我把物种这个术语看作是为了方便一组彼此非常相似的个体而任意给出的一个术语...它本质上与变体这个词没有什么不同,变体指的是不太明显和波动性更大的形式。同样,与单纯的个体差异相比,为了方便起见,多样性一词也是随意使用的。
我把物种这个术语看作是为了方便而任意赋予一组彼此非常相似的个体的...它与多样性这个词没有本质上的区别,多样性是指不太明显和更易波动的形式。同样,为了方便起见,与个体差异相比,多样性一词也是任意使用的。[133]
参考文献
- [1]
^"Species Concepts". Scientific American. 20 April 2012. Archived from the original on 14 March 2017. Retrieved 14 March 2017..
- [2]
^Mallet, James (1995). "A species definition for the modern synthesis". Trends in Ecology & Evolution. 10 (7): 294–299. doi:10.1016/0169-5347(95)90031-4..
- [3]
^Masters, J. C.; Spencer, H. G. (1989). "Why We Need a New Genetic Species Concept". Systematic Zoology. 38 (3): 270–279. doi:10.2307/2992287. JSTOR 2992287..
- [4]
^Mayden, R. L. (1997). Claridge, M. F.; Dawah, H. A.; Wilson, M. R., eds. A hierarchy of species concepts: the denouement of the species problem. The Units of Biodiversity – Species in Practice Special Volume 54. Systematics Association..
- [5]
^Zachos 2016, p. 79..
- [6]
^Gould, Stephen Jay (1980). "A Quahog is a Quahog". In: The Panda's Thumb: More Reflections in Natural History. New York: W.W. Norton & Company. pp. 204–213. ISBN 978-0-393-30023-9..
- [7]
^Maynard Smith, John (1989). Evolutionary Genetics. Oxford University Press. pp. 273–274. ISBN 978-0-19-854215-5..
- [8]
^Ruse, Michael (1969). "Definitions of Species in Biology". The British Journal for the Philosophy of Science. 20 (2): 97–119. doi:10.1093/bjps/20.2.97. JSTOR 686173..
- [9]
^Lewin, Ralph A. (1981). "Three Species Concepts". Taxon. 30 (3): 609–613. doi:10.2307/1219942. JSTOR 1219942..
- [10]
^Claridge et al.:404..
- [11]
^Ghiselin, Michael T. (1974). "A Radical Solution to the Species Problem". Systematic Biology. 23 (4): 536–544. doi:10.1093/sysbio/23.4.536..
- [12]
^Claridge et al.:408–409..
- [13]
^Paterson, H. E. H. (1985). Vrba, E. S., ed. Monograph No. 4: The recognition concept of species. Species and Speciation. Pretoria: Transvaal Museum..
- [14]
^Mallet, James. Calow, P., ed. Species, Concepts of (PDF). Encyclopaedia of Ecology and Environmental Management. Blackwell. pp. 709–711. ISBN 978-0-632-05546-3. Archived (PDF) from the original on 19 October 2016..
- [15]
^Kull, Kalevi (2016). "The biosemiotic concept of the species". Biosemiotics. 9: 61–71. doi:10.1007/s12304-016-9259-2. Archived from the original on 7 February 2018..
- [16]
^Stackebrandt, E.; Goebel, B. M. (1994). "Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology". Int. J. Syst. Bacteriol. 44 (4): 846–849. doi:10.1099/00207713-44-4-846..
- [17]
^Stackebrandt, E.; Ebers, J. (2006). "Taxonomic parameters revisited: tarnished gold standards" (PDF). Microbiology Today. 33 (4): 152–155. Archived (PDF) from the original on 25 March 2018..
- [18]
^Newton, Ian (2003). Speciation and Biogeography of Birds. Academic Press. p. 69. ISBN 978-0-08-092499-1. Archived from the original on 7 February 2018..
- [19]
^Andersson, Malte (1999). Hybridization and skua phylogeny. Proceedings of the Royal Society B. 266. pp. 1579–1585. doi:10.1098/rspb.1999.0818. ISBN 9780080924991. PMC 1690163. Archived from the original on 7 February 2018..
- [20]
^Keswani, J.; Whitman, W. B. (2001). "Relationship of 16S rRNA sequence similarity to DNA hybridization in prokaryotes". International Journal of Systematic and Evolutionary Microbiology. 51 (2): 667–78. doi:10.1099/00207713-51-2-667. PMID 11321113..
- [21]
^"What Is DNA Barcoding?". Barcode of Life. Archived from the original on 1 July 2017. Retrieved 11 October 2017..
- [22]
^Ratnasingham, Sujeevan; Hebert, Paul D. N. (2007). "BOLD: The Barcode of Life Data System (http://www.barcodinglife.org)". Molecular Ecology Notes. 7 (3): 355–364. doi:10.1111/j.1471-8286.2007.01678.x. PMC 1890991. PMID 18784790..
- [23]
^Stoeckle, Mark (November–December 2013). "DNA Barcoding Ready for Breakout". GeneWatch. 26 (5)..
- [24]
^DeSalle, R.; Egan, M. G.; Siddall, M. (2005). "The unholy trinity: taxonomy, species delimitation and DNA barcoding". Philosophical Transactions of the Royal Society B: Biological Sciences. 360 (1462): 1905–1916. doi:10.1098/rstb.2005.1722. PMC 1609226. PMID 16214748..
- [25]
^Whitworth, T. L.; Dawson, R. D.; Magalon, H.; Baudry, E. (2007). "DNA barcoding cannot reliably identify species of the blowfly genus Protocalliphora (Diptera: Calliphoridae)". Proceedings of the Royal Society B: Biological Sciences. 274 (1619): 1731–9. doi:10.1098/rspb.2007.0062. PMC 2493573. PMID 17472911..
- [26]
^Wheeler, Quentin D.; Platnick, Norman I. 2000. The phylogenetic species concept (sensu Wheeler & Platnick). In: Wheeler, Q. D.; Meier, R., editors. Species concepts and phylogenetic theory: a debate. New York: Columbia University Press. pp. 55–69..
- [27]
^Giraud, T.; Refrégier, G.; Le Gac, M.; de Vienne, D. M.; Hood, M. E. (2008). "Speciation in Fungi". Fungal Genetics and Biology. 45 (6): 791–802. doi:10.1016/j.fgb.2008.02.001. PMID 18346919..
- [28]
^Bernardo, J. (2011). "A critical appraisal of the meaning and diagnosability of cryptic evolutionary diversity, and its implications for conservation in the face of climate change". In Hodkinson, T.; Jones, M.; Waldren, S.; Parnell, J. Climate Change, Ecology and Systematics. Systematics Association Special Series. Cambridge University Press. pp. 380–438. ISBN 978-0-521-76609-8...
- [29]
^Nixon, K. C.; Wheeler, Q. D. (1990). "An amplification of the phylogenetic species concept". Cladistics. 6 (3): 211–223. doi:10.1111/j.1096-0031.1990.tb00541.x..
- [30]
^Taylor, J. W.; Jacobson, D. J.; Kroken, S.; Kasuga, T.; Geiser, D. M.; Hibbett, D. S.; Fisher, M. C. (2000). "Phylogenetic species recognition and species concepts in fungi". Fungal Genetics and Biology. 31 (1): 21–32. doi:10.1006/fgbi.2000.1228. PMID 11118132..
- [31]
^Taylor, J. W.; Turner, E.; Townsend, J. P.; Dettman, J. R.; Jacobson, D. (2006). "Eukaryotic microbes, species recognition and the geographic limits of species: Examples from the kingdom Fungi". Philosophical Transactions of the Royal Society B: Biological Sciences. 361 (1475): 1947–1963. doi:10.1098/rstb.2006.1923. PMC 1764934. PMID 17062413..
- [32]
^Zachos 2016, pp. 91–92..
- [33]
^Groves, C.; Grubb, P. 2011. Ungulate taxonomy. Baltimore, MD: The Johns Hopkins University Press..
- [34]
^Heller, R.; Frandsen, P.; Lorenzen, E.D.; Siegismund, H.R. (2013). "Are there really twice as many bovid species as we thought?". Systematic Biology. 62 (3): 490–493. doi:10.1093/sysbio/syt004. PMID 23362112..
- [35]
^Cotterill, F.; Taylor, P.; Gippoliti, S.; et al. (2014). "Why one century of phenetics is enough: Response to 'are there really twice as many bovid species as we thought?'". Systematic Biology. 63 (5): 819–832. doi:10.1093/sysbio/syu003. PMID 24415680..
- [36]
^Laporte, L. O. F. (1994). "Simpson on species". Journal of the History of Biology. 27 (1): 141–159. doi:10.1007/BF01058629. PMID 11639257..
- [37]
^Wheeler, pp. 70–92, 146–160, 198–208.
- [38]
^Ridley, Mark. "The Idea of Species". Evolution (2nd ed.). Blackwell Science. p. 719. ISBN 978-0-86542-495-1..
- [39]
^Baker, Robert J.; Bradley, Robert D. (2006). "Speciation in Mammals and the Genetic Species Concept". Journal of Mammalogy. 87 (4): 643–662. doi:10.1644/06-MAMM-F-038R2.1. PMC 2771874. PMID 19890476..
- [40]
^Government of Canada. Committee on the Status of Endangered Wildlife in Canada. "COSEWIC's Assessment Process and Criteria". Cosepac.gc.ca. Archived from the original on 12 April 2015. Retrieved 7 April 2015..
- [41]
^DeWeerdt, Sarah (29 July 2002). "What Really is an Evolutionarily Significant Unit?". University of Washington. Archived from the original on 5 February 2017. Retrieved 1 December 2016..
- [42]
^"Chronospecies". Oxford Reference. Archived from the original on 1 October 2014. Retrieved 12 January 2018..
- [43]
^Carr, Steven M. (2005). "Evolutionary species and chronospecies". Memorial University Newfoundland and Labrador. Archived from the original on 10 March 2016. Retrieved 12 January 2018..
- [44]
^Dzik, J. (1985). "Typologic versus population concepts of chronospecies: implications for ammonite biostratigraphy" (PDF). Acta Palaeontologica Polonica. 30 (1–2): 71–92. Archived (PDF) from the original on 13 March 2017..
- [45]
^O'Brien, Michael J.; Lyman, R. Lee (2007). Applying Evolutionary Archaeology: A Systematic Approach. Springer. pp. 146–149. ISBN 978-0-306-47468-2. Archived from the original on 7 February 2018..
- [46]
^Van Regenmortel, Marc H. V. (2010). "Logical puzzles and scientific controversies: The nature of species, viruses and living organisms". Systematic and Applied Microbiology. 33 (1): 1–6. doi:10.1016/j.syapm.2009.11.001. PMID 20005655..
- [47]
^van Nimwegen, Erik; Crutchfield, James P.; Huynen, Martijn (August 1999). "Neutral evolution of mutational robustness". PNAS. 96 (17): 9716–9720. arXiv:adap-org/9903006. Bibcode:1999PNAS...96.9716V. doi:10.1073/pnas.96.17.9716..
- [48]
^Wilke, Claus O.; Wang, Jia Lan; Ofria, Charles; Lenski, Richard E.; Adami, Christoph (2001). "Evolution of digital organisms at high mutation rates leads to survival of the flattest" (PDF). Nature. 412 (6844): 331–333. doi:10.1038/35085569. PMID 11460163..
- [49]
^Elena, S.F.; Agudelo-Romero, P.; Carrasco, P.; Codoñer, F.M.; Martín, S.; Torres-Barceló, C.; Sanjuán, R. (2008). "Experimental evolution of plant RNA viruses". Heredity. 100 (5): 478–483. doi:10.1038/sj.hdy.6801088. PMID 18253158..
- [50]
^"A Word About Species Names ..." Smithsonian Marine Station at Fort Pierce. Archived from the original on 24 March 2017. Retrieved 11 March 2017..
- [51]
^Hone, Dave (19 June 2013). "What's in a name? Why scientific names are important". The Guardian. Archived from the original on 15 February 2017. Retrieved 11 March 2017..
- [52]
^One example of an abstract of an article naming a new species can be found at Wellner, S.; Lodders, N.; Kämpfer, P. (2012). "Methylobacterium cerastii sp. nov., a novel species isolated from the leaf surface of Cerastium holosteoides". International Journal of Systematic and Evolutionary Microbiology. 62 (Pt 4): 917–924. doi:10.1099/ijs.0.030767-0. PMID 21669927. Retrieved 18 June 2011..
- [53]
^Hitchcock, A. S. (1921), "The Type Concept in Systematic Botany", American Journal of Botany, 8 (5): 251–255, doi:10.2307/2434993, JSTOR 2434993.
- [54]
^Nicholson, Dan H. "Botanical nomenclature, types, & standard reference works". Smithsonian National Museum of Natural History, Department of Botany. Archived from the original on 16 September 2015. Retrieved 17 November 2015..
- [55]
^"International Code of Zoological Nomenclature, Recommendation 25C". Archived from the original on 8 June 2011. Retrieved 18 June 2011..
- [56]
^Winston, Judith E. (1999). Describing species. Practical taxonomic procedure for biologists. New York: Columbia University Press. pp. 141–144..
- [57]
^"Home – Taxonomy – NCBI". Ncbi.nlm.nih.gov. 19 October 2012. Archived from the original on 7 February 2018. Retrieved 25 November 2012..
- [58]
^"KEGG Organisms: Complete Genomes". Genome.jp. Archived from the original on 14 October 2012. Retrieved 25 November 2012..
- [59]
^"Taxonomy". Uniprot.org. Archived from the original on 27 November 2012. Retrieved 25 November 2012..
- [60]
^"ITIS: Homo sapiens". Catalogue of Life. Archived from the original on 12 March 2017. Retrieved 11 March 2017..
- [61]
^Simpson, George Gaylord (1945). "The Principles of Classification and a Classification of Mammals". Bulletin of the American Museum of Natural History. 85: 23..
- [62]
^Chase, Bob (2005). "Upstart Antichrist". History Workshop Journal (60): 202–206. doi:10.1093/hwj/dbi042..
- [63]
^Wilson, Philip (2016). "sensu stricto, sensu lato". AZ of tree terms. Archived from the original on 10 January 2018. Retrieved 9 January 2018..
- [64]
^"Glossary: sensu". International Commission on Zoological Nomenclature. Archived from the original on 2 August 2017. Retrieved 9 January 2018..
- [65]
^Mayr, Ernst (1942). Systematics and the Origin of Species. New York: Columbia University Press..
- [66]
^Wheeler, pp. 17–29.
- [67]
^Hopf, F. A.; Hopf, F. W. (1985). "The role of the Allee effect on species packing". Theoretical Population Biology. 27: 27–50. doi:10.1016/0040-5809(85)90014-0..
- [68]
^Bernstein, H.; Byerly, H. C.; Hopf, F. A.; Michod, R.E. (1985). "Sex and the emergence of species". Journal of Theoretical Biology. 117 (4): 665–690. doi:10.1016/S0022-5193(85)80246-0. PMID 4094459..
- [69]
^Bernstein, Carol; Bernstein, Harris (1991). Aging, sex, and DNA repair. Boston: Academic Press. ISBN 978-0-12-092860-6..
- [70]
^Michod, Richard E. (1995). Eros and Evolution: A Natural Philosophy of Sex. Addison-Wesley. ISBN 978-0-201-44232-8..
- [71]
^Hanage, William P. (2013), "Fuzzy species revisited", BMC Biology, 11 (41): 41, doi:10.1186/1741-7007-11-41, PMC 3626887, PMID 23587266.
- [72]
^de Queiroz, K. (2005). "Ernst Mayr and the modern concept of species". PNAS. 102 (Supplement 1): 6600–6607. Bibcode:2005PNAS..102.6600D. doi:10.1073/pnas.0502030102. PMC 1131873. PMID 15851674..
- [73]
^De Queiroz, K. (2007). "Species concepts and species delimitation". Systematic Biology. 56 (6): 879–886. doi:10.1080/10635150701701083. PMID 18027281..
- [74]
^Fraser, C.; Alm, E. J.; Polz, M. F.; Spratt, B. G.; Hanage, W. P. (2009). "The bacterial species challenge: making sense of genetic and ecological diversity". Science. 323 (5915): 741–746. Bibcode:2009Sci...323..741F. doi:10.1126/science.1159388. PMID 19197054..
- [75]
^Gevers, Dirk; Cohan, Frederick M.; Lawrence, Jeffrey G.; Spratt, Brian G.; Coenye, Tom; Feil, Edward J.; Stackebrandt, Erko; De Peer, Yves Van; Vandamme, Peter; Thompson, Fabiano L.; Swings, Jean (2005). "Opinion: Re-evaluating prokaryotic species". Nature Reviews Microbiology. 3 (9): 733–9. doi:10.1038/nrmicro1236. PMID 16138101..
- [76]
^Templeton, A. R. (1989). "The meaning of species and speciation: A genetic perspective". In Otte, D.; Endler, J. A. Speciation and its consequences. Sinauer Associates. pp. 3–27..
- [77]
^Edward G. Reekie; Fakhri A. Bazzaz (2005). Reproductive allocation in plants. Academic Press. p. 99. ISBN 978-0-12-088386-8. Archived from the original on 17 June 2013..
- [78]
^Rosselló-Mora, Ramon; Amann, Rudolf (January 2001). "The species concept for prokaryotes". FEMS Microbiology Reviews. 25 (1): 39–67. doi:10.1111/j.1574-6976.2001.tb00571.x. PMID 11152940..
- [79]
^Andino, Raul; Domingo, Esteban (2015). "Viral quasispecies". Virology. 479–480: 46–51. doi:10.1016/j.virol.2015.03.022. PMC 4826558. PMID 25824477..
- [80]
^Biebricher, C. K.; Eigen, M. (2006). Quasispecies: Concept and Implications for Virology. Current Topics in Microbiology and Immunology. Springer. pp. 1–31. doi:10.1007/3-540-26397-7_1. ISBN 978-3-540-26397-5..
- [81]
^Teueman, A. E. (2009). "The Species-Concept in Palaeontology". Geological Magazine. 61 (8): 355–360. Bibcode:1924GeoM...61..355T. doi:10.1017/S001675680008660X. Archived from the original on 14 March 2017..
- [82]
^Zachos 2016, p. 101..
- [83]
^Zachos 2016, pp. 156–157..
- [84]
^Lahr, D.J.; Laughinghouse, H.D.; Oliverio, A. M.; Gao, F.; Katz, L. A. (2014). "How discordant morphological and molecular evolution among microorganisms can revise our notions of biodiversity on Earth". BioEssays. 36 (10): 950–959. doi:10.1002/bies.201400056. PMC 4288574. PMID 25156897..
- [85]
^Bapteste, E.; et al. (May 2005). "Do orthologous gene phylogenies really support tree-thinking?". BMC Evolutionary Biology. 5 (33): 33. doi:10.1186/1471-2148-5-33. PMC 1156881. PMID 15913459..
- [86]
^Williamson, David I. (2003). The Origins of Larvae. Kluwer. ISBN 978-1-4020-1514-4..
- [87]
^Heywood, V. H. (1962). "The 'species aggregate' in theory and practice". In Heywood, V. H.; Löve, Á. Symposium on Biosystematics, Montreal, October 1962. pp. 26–36..
- [88]
^Pimentel, David (2014). Biological Invasions: Economic and Environmental Costs of Alien Plant, Animal, and Microbe Species. CRC Press. p. 92. ISBN 978-1-4200-4166-8. Archived from the original on 7 February 2018..
- [89]
^Jarvis, C. E. (1992). "Seventy-Two Proposals for the Conservation of Types of Selected Linnaean Generic Names, the Report of Subcommittee 3C on the Lectotypification of Linnaean Generic Names". Taxon. 41 (3): 552–583. doi:10.2307/1222833. JSTOR 1222833..
- [90]
^Wittzell, Hakan (1999). "Chloroplast DNA variation and reticulate evolution in sexual and apomictic sections of dandelions". Molecular Ecology. 8 (12): 2023–2035. doi:10.1046/j.1365-294x.1999.00807.x. PMID 10632854..
- [91]
^Dijk, Peter J. van (2003). "Ecological and evolutionary opportunities of apomixis: insights from Taraxacum and Chondrilla". Philosophical Transactions of the Royal Society B. 358 (1434): 1113–1121. doi:10.1098/rstb.2003.1302. PMC 1693208. PMID 12831477..
- [92]
^Mallet, James; Beltrán, M.; Neukirchen, W.; Linares, M. (2007). "Natural hybridization in heliconiine butterflies: the species boundary as a continuum". BMC Evolutionary Biology. 7 (1): 28. doi:10.1186/1471-2148-7-28. PMC 1821009. PMID 17319954..
- [93]
^Ron, Santiago; Caminer, Marcel (2014). "Systematics of treefrogs of the Hypsiboas calcaratus and Hypsiboas fasciatus species complex (Anura, Hylidae) with the description of four new species". ZooKeys (370): 1–68. doi:10.3897/zookeys.370.6291. PMC 3904076. PMID 24478591..
- [94]
^Geml, J.; Tulloss, R. E.; Laursen, G. A.; Sasanova, N. A.; Taylor, D. L. (2008). "Evidence for strong inter- and intracontinental phylogeographic structure in Amanita muscaria, a wind-dispersed ectomycorrhizal basidiomycete". Molecular Phylogenetics and Evolution. 48 (2): 694–701. doi:10.1016/j.ympev.2008.04.029. PMID 18547823..
- [95]
^"Defining a species". University of California Berkeley. Archived from the original on 13 March 2017. Retrieved 12 March 2017..
- [96]
^Zachos 2016, p. 188..
- [97]
^Stamos, David N. (2003). The Species Problem: Biological Species, Ontology, and the Metaphysics of Biology. Lexington Books. p. 330. ISBN 978-0-7391-6118-0. Archived from the original on 12 March 2017..
- [98]
^Moritz, C.; Schneider, C. J.; Wake, D. B. (1992). "Evolutionary Relationships Within the Ensatina Eschscholtzii Complex Confirm the Ring Species Interpretation" (PDF). Systematic Biology. 41 (3): 273. doi:10.1093/sysbio/41.3.273. Archived (PDF) from the original on 12 January 2018..
- [99]
^Irwin, D. E.; Bensch, Staffan; Irwin, Jessica H.; Price, Trevor D. (2005). "Speciation by Distance in a Ring Species". Science. 307 (5708): 414–6. Bibcode:2005Sci...307..414I. doi:10.1126/science.1105201. PMID 15662011..
- [100]
^Martens, Jochen; Päckert, Martin (2007). "Ring species – Do they exist in birds?". Zoologischer Anzeiger. 246 (4): 315. doi:10.1016/j.jcz.2007.07.004..
- [101]
^Alcaide, M.; Scordato, E. S. C.; Price, T. D.; Irwin, D. E. (2014). "Genomic divergence in a ring species complex". Nature. 511 (7507): 83–85. Bibcode:2014Natur.511...83A. doi:10.1038/nature13285. hdl:10261/101651. PMID 24870239..
- [102]
^Liebers, Dorit; Knijff, Peter de; Helbig, Andreas J. (2004). "The herring gull complex is not a ring species". Proc Biol Sci. 271 (1542): 893–901. doi:10.1098/rspb.2004.2679. PMC 1691675. PMID 15255043..
- [103]
^Highton, R. (1998). "Is Ensatina eschscholtzii a ring species?". Herpetologica. 54 (2): 254–278. JSTOR 3893431..
- [104]
^Barton, N. H. (June 2010). "What role does natural selection play in speciation?". Philosophical Transactions of the Royal Society B. 365 (1547): 1825–1840. doi:10.1098/rstb.2010.0001. PMC 2871892. PMID 20439284..
- [105]
^Vaux, Felix; Trewick, Steven A.; Morgan-Richards, Mary (2017). "Speciation through the looking-glass". Biological Journal of the Linnean Society. 120 (2): 480–488. doi:10.1111/bij.12872..
- [106]
^Zachos 2016, pp. 77–96..
- [107]
^Cook, Orator F. (30 March 1906). "Factors of species-formation". Science. 23 (587): 506–507. Bibcode:1906Sci....23..506C. doi:10.1126/science.23.587.506. PMID 17789700..
- [108]
^Cook, Orator F. (November 1908). "Evolution Without Isolation". The American Naturalist. 42 (503): 727–731. doi:10.1086/279001..
- [109]
^Via, Sara (16 June 2009). "Natural selection in action during speciation". Proc. Natl. Acad. Sci. U.S.A. 106 (Suppl 1): 9939–9946. Bibcode:2009PNAS..106.9939V. doi:10.1073/pnas.0901397106. PMC 2702801. PMID 19528641..
- [110]
^Mayr, Ernst (1982). "Speciation and Macroevolution". Evolution. 36 (6): 1119–1132. doi:10.1111/j.1558-5646.1982.tb05483.x. PMID 28563569..
- [111]
^Pennisi, Elizabeth (2004). "Researchers Trade Insights about Gene Swapping" (PDF). Science. 334–335: 335. Archived from the original (PDF) on 18 February 2006..
- [112]
^Melcher, Ulrich (2001). "Molecular genetics: Horizontal gene transfer". Oklahoma State University. Archived from the original on 4 March 2016..
- [113]
^Venton, Danielle (2017). "Highlight: Applying the Biological Species Concept across All of Life". Genome Biology and Evolution. 9 (3): 502–503. doi:10.1093/gbe/evx045. PMC 5381533. PMID 28391326..
- [114]
^Bobay, Louis-Marie; Ochman, Howard (2017). "Biological Species Are Universal across Life's Domains". Genome Biology and Evolution. 9 (3): 491–501. doi:10.1093/gbe/evx026. PMC 5381558. PMID 28186559..
- [115]
^Kunin, W. E.; Gaston, Kevin, eds. (1996). The Biology of Rarity: Causes and consequences of rare–common differences. ISBN 978-0-412-63380-5. Archived from the original on 5 September 2015..
- [116]
^Stearns, Beverly Peterson; Stearns, Stephen C. (2000). Watching, from the Edge of Extinction. New Haven, London: Yale University Press. p. preface x. ISBN 978-0-300-08469-6..
- [117]
^Zachos 2016, p. 82..
- [118]
^Zachos, Frank E. (2015). "Taxonomic inflation, the Phylogenetic Species Concept and lineages in the Tree of Life – a cautionary comment on species splitting". Journal of Zoological Systematics and Evolutionary Research. 53 (2): 180–184. doi:10.1111/jzs.12088..
- [119]
^Agapow, Paul‐Michael; Bininda‐Emonds, Olaf R. P.; Crandall, Keith A.; Gittleman, John L.; Mace, Georgina M.; Marshall, Jonathon C.; Purvis, Andy (2004). "The Impact of Species Concept on Biodiversity Studies" (PDF). The Quarterly Review of Biology. 79 (2): 161–179. CiteSeerX 10.1.1.535.2974. doi:10.1086/383542. JSTOR 10.1086/383542. Archived (PDF) from the original on 11 January 2018..
- [120]
^Hey, Jody (July 2001). "The mind of the species problem". Trends in Ecology and Evolution. 16 (7): 326–329. doi:10.1016/S0169-5347(01)02145-0. PMID 11403864..
- [121]
^Haig, Susan M.; Allendorf, F.W. (2006). "Hybrids and Policy". In Scott, J. Michael; Goble, D. D.; Davis, Frank W. The Endangered Species Act at Thirty, Volume 2: Conserving Biodiversity in Human-Dominated Landscapes. Washington: Island Press. pp. 150–163. Archived from the original on 7 February 2018..
- [122]
^Mishler, Brent D. (1999). Wilson, R., ed. Getting Rid of Species? (PDF). Species: New Interdisciplinary Essays. MIT Press. pp. 307–315. ISBN 978-0262731232. Archived (PDF) from the original on 10 January 2018..
- [123]
^Vane-Wright, R. I.; Humphries, C. J.; Williams, P. H. (1991). "What to protect? – systematics and the agony of choice". Biological Conservation. 55 (3): 235–254. doi:10.1016/0006-3207(91)90030-D..
- [124]
^Leroi, Armand Marie (2014). The Lagoon: How Aristotle Invented Science. Bloomsbury. pp. 88–90. ISBN 978-1-4088-3622-4..
- [125]
^Ray, John (1686). Historia plantarum generalis, Tome I, Libr. I. p. Chap. XX, page 40., quoted in Mayr, Ernst (1982). The growth of biological thought: diversity, evolution, and inheritance. Belknap Press. p. 256..
- [126]
^Davis, P. H.; Heywood, V. H. (1973). Principles of Angiosperm Taxonomy. Huntington, New York: Robert E. Krieger Publishing Company. p. 17..
- [127]
^Reveal, James L.; Pringle, James S. (1993). "7. Taxonomic Botany and Floristics". Flora of North America. Oxford University Press. pp. 160–161. ISBN 978-0-19-505713-3..
- [128]
^Simpson, George Gaylord (1961). Principles of Animal Taxonomy. Columbia University Press. pp. 56–57..
- [129]
^Mahoney, Edward P. (1987). "Lovejoy and the Hierarchy of Being". Journal of the History of Ideas. 48 (2): 211–230. doi:10.2307/2709555. JSTOR 2709555..
- [130]
^"Carl Linnaeus (1707–1778)". UCMP Berkeley. Archived from the original on 30 April 2011. Retrieved 24 January 2018..
- [131]
^Gould, Stephen Jay (2002). The Structure of Evolutionary Theory. Harvard: Belknap Harvard. pp. 170–197. ISBN 978-0-674-00613-3..
- [132]
^Bowler, Peter J. (2003). Evolution: The History of an Idea (3rd ed.). Berkeley, CA: University of California Press. pp. 177–223 and passim. ISBN 978-0-520-23693-6..
- [133]
^Menand, Louis (2001). The Metaphysical Club: A Story of Ideas in America. Farrar, Straus and Giroux. pp. 123–124. ISBN 978-0-374-70638-8..
暂无