基因线路
编辑基因线路(Genetic circuit)是一个合成生物学中常见的概念,用于统称生物体内广泛存在的,用于控制各种各样生命活动的动态调控系统。生物体内动态调控系统并不是一个合成生物学的新概念,而是生物学的传统研究领域。从已经进入生物学可能多年的,细菌中经典的乳糖操纵子和色氨酸操纵子,到获得2019年诺贝尔生理学和医学奖的氧信号通路,都是对生物体内方方面面活动进行控制的机制。动态调控系统与基因线路概念的区别在于:动态调控系统是传统生物学领域对生命中各种控制系统的统称,对其研究目的一般是为了对各种生命过程进行深入了解,产出的是对生命本质更深刻的认知;而基因线路是一个存在于合成生物学领域的概念,相比对生命的认知,更加强调对知识的利用,实现需求导向的设计,更加具有工程化的属性。
1 合成生物学的基本概念编辑
为了准确理解基因线路的概念,需要对合成生物学有基本的了解。合成生物学区别于传统生物学的思想在于对工程设计原理的系统性应用,目的从认识自然进化到到改造自然,设计自然,终极目标是人造生命。
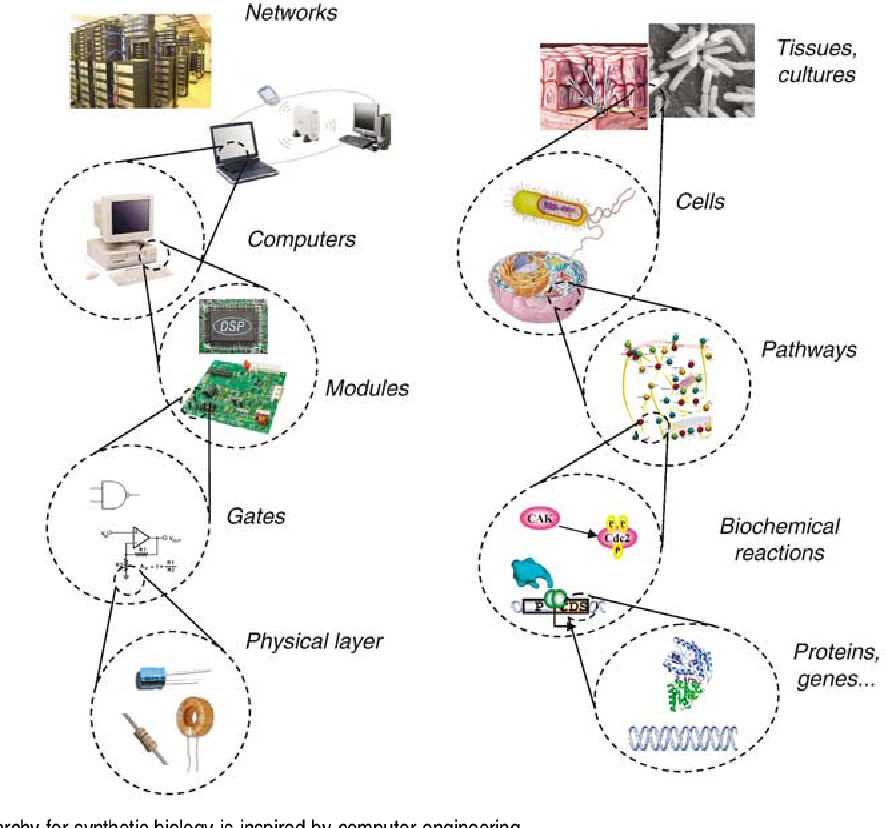
所谓人造生命,就是使用简单的元件,从底层到高层,从简单到复杂的构建出的生命体。可以粗略的与一台计算机进行类比[1]。人造生命中底层中具有特定功能的蛋白质,核酸等元件可类比为电路中底层的二级管,电阻等元件;由这些基本生物元件以适当方式组成的功能模块可以实现类似于电子芯片中逻辑门的功能,如“与”、“或”、“非”等布尔逻辑门;通过多种逻辑门的组合,可以搭建具有复杂功能的调控网络,对特定输入信号进行判断并输出信号,类似于完整的计算机芯片;最后,由多种复杂网络集合,组成能够实现自我代谢,繁殖的细胞或单细胞生命,就是人造生命体,类比于由多种芯片、电路组成的计算机;而在此基础上,由单细胞搭建的组织,就像互联网一样——由许多计算机组成并相互连接,实现更复杂的功能。
从以上类比可以看出,合成生物学中的概念,与电子学很有渊源,借鉴了很多电子学的称谓,如线路(circuit)、元件(part)、逻辑门(logic gate)、网络(network)等。而基因线路,就是类似于电路(electrical circuit)的概念,是各种元件的集合,是实现复杂功能的载体。
2 自然界中存在的基因线路编辑
自然界中存在大量的天然基因线路,是生命体用于收集和处理各种体内生化信号和体外环境变化的动态调控系统,与物质代谢、能量供给共同构成了生命过程的三个基本要素。所有生物体均由物质构成,由能量驱动,由基因线路控制物质的代谢和能量的流动,以实现多样的生理活动,如细胞分裂,个体形态发育等。同时,生物体对外界环境变化的适应和抗逆反应也依赖于基因线路,如人体细胞在血糖浓度升高时对胰岛素的分泌,组织受损时启动组织再生,大量失血时促进造血干细胞分化等。[2]
3 人工基因线路编辑
人工基因线路是对天然基因调控线路进行的简单化处理和重新编程,以及引入自然界不存在的人造法则。
与天然基因线路不同,人工基因线路存在的目的是根据人工生命体的设计目标,进行有针对性的控制,实现特定的控制逻辑,发挥类似于计算机控制芯片的功能。因此,人工基因线路是对生命进行可编程控制的具体体现,是合成生物学的标志性技术。对生命的编程如同电路编程一样,需要使用大量工程化的元器件,如计数器、脉冲信号产生器、逻辑信号门、信号过滤器等[3][4][5][6][7][8],以实现从低级到高级,由简单到复杂的控制。为了构建出此类元器件,需要在传统生物学多年来对生命调控法则的认识基础上,按照工程化设计原理对生命系统进行简单化处理,再进一步按照不同的方式进行组合而成。尽管人工基因线路与电子线路都是信号采集和处理的信息运算系统,两者在很多方面又是截然不同的。基因线路的工作环境是动态生长的活体细胞,是大量各种分子的混合物,而电子线路的工作环境是固体金属和半导体材料,各个元件之间很容易实现绝缘。这些差异决定了针对基因线路的设计与组装必须要探索新途径,而不是简单地照搬照抄电子线路的成功方案。
4 人工基因线路的应用编辑
计算机芯片是各种电器中不可缺少的核心设备,控制着电器的各种功能,同时带来“智能化”的响应。与之类似,人工基因线路作为各种合成生物学应用的可编程控制组件,往往能够实现一些传统技术难以实现的、“智能化”的控制方式。
在肿瘤治疗领域,CAR-T技术已经展现出了有目共睹的治疗效果,但也在T细胞激活水平调节、靶向特异性、信号通路调控等方面存在提升的空间。针对这些问题,波士顿大学的Wong课题组提出了一种新型的CAR-T设计方案——“SUPRA CAR”,被诸多科技媒体称为新一代的CAR-T疗法,引起了广泛关注[9]。SUPRA CAR方案通过将CAR-T固定式的细胞外scFV单链抗体,细胞内CD3z信号结构域拆分为由亮氨酸拉链(Leucine-Zipper)这种通用结构连接的两部分,分别实现了对两部分的模块化设计和可编程性,为人工基因线路设计创造了可能。基于该方案设计的人工基因线路能够对多种抗原信号产生逻辑响应,并且调控不同免疫细胞类型的信号通路。通过合适的线路设计,还实现了调节T细胞激活反应强度,以减轻治疗的副作用。
此外,在代谢工程领域,人工基因线路也展现出了“智能化”控制的潜力,提供了更好的解决方案。基于细菌群体感应功能设计的人工基因线路能够根据细菌数量对目的基因表达进行动态调节,使对细菌生长有负面影响的基因在细菌达到一定数量之后再表达,规避了传统发酵过程中微生物生长和发酵产物生产的矛盾,实现了对发酵过程“先生长,再生产”的动态调控,同时也避免了使用昂贵的诱导剂[20]。
5 人工基因线路的挑战编辑
5.1 适应多种底盘生物
使用人工基因线路执行控制功能时,需要将该线路装载到不同的底盘生物中。不同领域的应用需要非常多样化的底盘生物,这就要求人工基因线路对底盘生物进行适应性。为了达到此目标,需要对人工基因线路进行“模块化”设计。“模块化”是合成生物学元件的核心属性之一,设计目标是将生物系统拆解为功能上相互独立的模块,并保证模块间的拼装不会导致模块功能的改变。模块化设计能使构建的生物系统像电子系统一样进行规模扩展和尺度放大,因此合成生物学领域的大量工作都注重模块化元件的开发。然而,细胞内的基因线路并不能像电子元件一样严格隔离,而是与细胞的生理状态耦合形成一个整体,人工基因线路会对细胞生理产生一些不可预知的干涉性影响,使基因线路本身不再“模块化”。这就导致在一种生物中精细刻画过的元件性质并不能在另一种生物中直接成立,因为元件一旦脱离了刻画时的细胞生理状态,其行为就有可能偏离预期。如果不能克服这个问题,合成生物元件就不能像电子元件一样使用简单元件逐步搭建复杂线路,而是需要耗费大量时间和精力对单个底盘生物中的元件进行点对点的优化。
5.2 细胞生长压力和细胞毒性
细胞利用有限的资源完成营养物质摄取、能量代谢、DNA复制、细胞分裂等诸多生理过程。为了优化自身的生长,细胞需要根据环境平衡分配这些资源。当人工基因线路的加入打破了这种平衡,就可能引发细胞生长压力(burden),影响细胞的正常生长。细胞的生长压力主要表现在两个方面:① 人工基因线路在蛋白表达过程中占用了底盘细胞的RNA聚合酶和核糖体,还有相关的辅酶,能量等资源。② 过量表达蛋白还可能引起细胞的应激反应,激活一些细胞应激途径如ppGpp等。除了生长压力,人工基因线路还可能带来细胞毒性(toxicity)。与生长压力不同,细胞毒性产生的原因是人工基因线路调控过程中的脱靶效应对底盘细胞的正常生理活动产生的干扰,而非由于对细胞内基础资源的占用。
许多已经广泛使用的优质元件也因为自身的细胞毒性限制了其发挥和推广。例如,CRISPRi-dCas9(CRISPR interference-dCas9)调控系统被认为是优秀的可编程的调控元件,目前已被广泛用于基因线路的构建。然而,随着dCas9表达量升高,细胞生长会出现明显的迟滞。一些研究者认为该毒性来源于CRISPR的脱靶效应。因而在CRISPRi-dCas9的实际使用过程中,研究者需要耗费大量精力来平衡转录调控开关的正面影响与其对细胞生长的负面影响。合成生物学的元件设计最终要针对下游应用问题进行调整,而下游应用需要宿主细胞健康、快速生长,因此元件的细胞毒性将是合成生物学走向应用的瓶颈问题之一。
参考文献
- [1]
^Ernesto Andrianantoandro, Subhayu Basu,David K Karig,Ron Weiss.《Synthetic biology: new engineering rules foran emerging discipline》.Molecular Systems Biolog,2006,
- [2]
^娄春波,杜沛,孟凡康,季翔宇,张益豪.《人工基因线路的研究进展和未来挑战》.中国科学院院刊,2018,
- [3]
^Elowitz M B, Leibler S..《A synthetic oscillatory network of transcriptional regulators.》.Nature,2000,
- [4]
^Gardner T S, Cantor C R, Collins J J.《Construction of a genetic toggle switch in Escherichia coli》.Nature,2000,
- [5]
^You L, Cox R S, 3rd, Weiss R.《Programmed population control by cell-cell communication and regulated killing》.Nature,2004,
- [6]
^Levskaya A, Chevalier A A, Tabor J J, et al..《Engineering Escherichia coli to see light - These smart bacteria 'photograph' a light pattern as a high-definition chemical image.》.Nature,2005,
- [7]
^Anderson J C, Clarke E J, Arkin A P, et al..《nvironmentally controlled invasion of cancer cells by engineered bacteria.》.Journal of Molecular Biology,2006,
- [8]
^Balagadde F K, Song H, Ozaki J, et al..《A synthetic Escherichia coli predator-prey ecosystem》.Molecular Systems Biolog,2008,
- [9]
^Cho J H, Collins J J, Wong W W.《Universal Chimeric Antigen Receptors for Multiplexed and Logical Control of T Cell Responses》.Cell,2018,
暂无