线粒体DNA
编辑线粒体脱氧核糖核酸(mtDNA或mDNA)[1]是位于真核细胞内线粒体、细胞器中的脱氧核糖核酸,能将食物中的化学能转化为细胞可以利用的形式——三磷酸腺苷(ATP)。线粒体DNA只是真核细胞中的一小部分;大多数的脱氧核糖核酸可以在细胞核中找到,在植物和藻类中,也可以在质体如叶绿体中找到。
在人体中,线粒体基因的16,569个碱基对只编码37个基因。人类线粒体DNA是人类基因组中第一个被测序的重要部分。在包括人类在内的大多数物种中,mtDNA通常只从母亲那里遗传[2][3]。然而,在特殊情况下,人类婴儿有时会从他们的父亲和母亲那里继承mtDNA,导致mtDNA的异源性[4]。
由于动物mtDNA进化得比核遗传标记更快[5][6][7],所以mtDNA代表了系统发育学和进化生物学的支柱。它还允许检查人口的关联性,因此在人类学和生物地理学中变得很重要。
目录编辑
1 起源编辑
细胞核和线粒体的脱氧核糖核酸被认为是不同的进化起源,mtDNA是从被当今真核细胞的早期祖先吞噬的细菌的环状基因组中衍生出来的。这个理论被称为内共生理论。每个线粒体估计包含2-10个mtDNA拷贝[8]。在现存生物体的细胞中,线粒体中存在的绝大多数蛋白质(哺乳动物中大约有1500种不同类型)是由细胞核脱氧核糖核酸编码的,但是其中一些(如果不是大多数的话)的基因被认为最初是细菌来源的,后来在进化过程中被转移到真核细胞核中。
线粒体保留一些基因的原因是有争议的。在一些缺乏基因组的线粒体衍生细胞器物种中的存在表明[9],完全基因丢失是可能的,将线粒体基因转移到细胞核有几个优点[10]。 将远程生产的疏水蛋白产品靶向线粒体的困难是为什么一些基因保留在mtDNA的一个假设[11];氧化还原调节的共定位是另一个例子,引用了对线粒体机制进行局部控制的可取性[12]。最近对大量mtDNA基因组的分析表明,这两个特征可能决定线粒体基因的保留。
2 线粒体遗传编辑
在大多数多细胞生物中,mtDNA是从母亲那里遗传的(母系遗传)。其机制包括简单稀释(一个卵子平均含有200,000个mtDNA分子,而据报道健康的人类精子平均含有5个分子)[13][14],精子mtDNA在男性生殖道和受精卵中的降解;而且,至少在一些生物体中,精子mtDNA不能进入卵子。不管是什么机制,这种mtDNA遗传的单亲(单亲遗传)模式存在于大多数动物、植物和真菌中。
2.1 母系遗传
在有性生殖中,线粒体通常只从母亲那里遗传;哺乳动物精子中的线粒体通常在受精后被卵细胞破坏。此外,大多数线粒体位于精子尾部的底部,用于推进精子细胞;有时在受精过程中尾巴会丢失。1999年,有报道称父系精子线粒体(含有mtDNA)被标记了泛素,以便于它们随后在胚胎内部破坏的选择[15]。一些体外受精技术,特别是将精子注入卵母细胞,可能会干扰这一过程。
线粒体DNA是母系遗传的这一事实使得系谱研究人员能够追溯到很久以前的母系谱系。(父代遗传的Y染色体脱氧核糖核酸以类似的方式用于确定父系历史。)这通常是通过对高变控制区(HVR1或HVR2)进行测序,有时对线粒体的完整分子进行测序,作为一种系谱测试,在人类线粒体上完成的[16]。 例如, HVR1由大约440个碱基对组成。将这440个碱基对与其他个体(特定人群或数据库中的受试者)的相同区域进行比较,以确定母体谱系。通常,与修订后的剑桥参考序列进行比较。Vilà等人发表了追踪家犬母系血统的研究[17]。线粒体夏娃的概念是基于同样类型的分析,试图通过及时追踪血统来发现人类的起源。
mtDNA是高度保守的,其相对较慢的突变率(与微卫星等其他脱氧核糖核酸区域相比)使其有助于研究生物的进化关系——系统发育。生物学家可以确定并比较不同物种之间的mtDNA序列,并利用这些比较为所研究的物种构建进化树。然而,由于突变率较低,通常很难在很大程度上区分密切相关的物种,因此必须使用其他分析方法。
2.2 线粒体瓶颈
受单亲遗传且几乎没有重组的实体可能会受到穆勒棘轮效应的影响,即有害突变的累积,直至功能丧失。动物线粒体群体通过被称为mtDNA瓶颈的发育过程来避免这种情况。瓶颈是利用细胞中的随机过程,随着生物体的发育,增加细胞间突变负荷的可变性:具有一定比例突变mtDNA的单个卵细胞因此产生不同细胞具有不同突变负荷的胚胎。细胞水平的选择可以去除那些具有更多突变mtDNA的细胞,导致世代间突变负荷的稳定或减少。瓶颈背后的机制是有争议的[18][19][20][21] ,最近的数学和实验元研究为细胞分裂时mtDNA的随机分配和细胞内mtDNA分子的随机周转提供了证据。
2.3 父系遗传
普利茅斯石鸡中发现了雄性线粒体DNA遗传[22]。 证据也支持一些哺乳动物雄性线粒体遗传的罕见例子。具体来说,在老鼠身上存在记录在案的事件[23][24],雄性遗传的线粒体随后被排斥。在绵羊[25]和克隆牛[26]中也发现了这种在, 克人类中罕见的已有记载的男性线粒体遗传的病例[27][28]。尽管其中许多病例涉及克隆胚胎或随后父本线粒体的排斥,但其他病例记录了在实验室条件下的体内遗传和持久性。
在双壳类软体动物中观察到mtDNA的双单亲遗传。在这些物种中,雌性只有一种F型的mtDNA,而雄性的体细胞中有F型的mtDNA,但生殖细胞中还有M型的mtDNA(差异可达30%)[29]。在一些昆虫中还报道了父系遗传的线粒体,如果蝇[30][31] 、蜜蜂[32]、和周期性蝉[33]。
2.4 线粒体捐赠
一种被称为线粒体捐献或线粒体替代疗法的体外受精技术,可以从捐献的女性身上获得含有mtDNA的后代,从父母身上获得核基因。在纺锤体转移过程中,卵子的细胞核被插入到捐献女性的细胞质中,捐献女性的细胞核被切除,但仍含有捐献女性的mtDNA。然后复合卵子与男性精子受精。当有遗传缺陷线粒体的女性希望生育和生产具有健康线粒体的后代时,就会使用这种方法[34]。第一个因线粒体捐赠而出生的已知儿童是一对约旦夫妇于2016年4月6日在墨西哥出生的男孩[35]。
3 结构编辑
3.1 环形与线性
在大多数多细胞生物中,mtDNA(或称有丝分裂基因组)被组织成环状、共价闭合的双链脱氧核糖核酸。但是在许多单细胞生物体中(例如纤毛虫四膜虫和绿藻莱茵衣藻),在极少数情况下也在多细胞生物体中(例如在某些刺胞藻属物种中),mtDNA被发现为线性组织的脱氧核糖核酸。大多数线性线粒体DNA拥有不同复制模式的端粒酶非依赖性端粒(即线性脱氧核糖核酸的末端),这使它们成为有趣的研究对象,因为许多具有线性mtDNA的单细胞生物是已知的病原体[36]。
3.2 哺乳动物
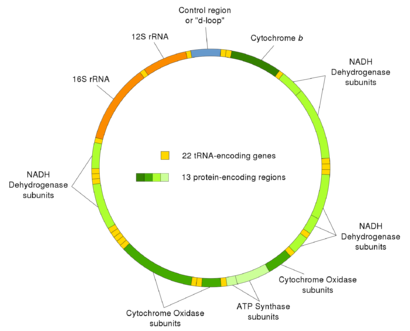
在人类线粒体的脱氧核糖核酸中(可能在一般后生动物中),每个体细胞通常有100-10,000个独立的mtDNA拷贝。在哺乳动物中,每个双链环状mtDNA分子由15,000-17,000个碱基对组成[37] 。mtDNA的两条链通过它们的核苷酸含量来区分,富含鸟嘌呤的链称为重链(或称H链),富含胞嘧啶的链称为轻链(或称L链)。然而,这些链的标记混乱是普遍的;这种混乱似乎源于1999年一篇有影响力的文章中将大多数编码链确定为重链[38]。轻链编码28个基因,重链编码9个基因,总共37个基因[39]。在这37个基因中,13个用于蛋白质(多肽),22个用于转移核糖核酸,两个用于核糖体核糖核酸的大小亚基[39]。人类线粒体基因组包含重叠基因(ATP8和ATP6以及ND4L和ND4),这是动物核基因组中罕见的特征。37基因模式也见于大多数后生动物,尽管mtDNA体型范围更大,在某些情况下,这些基因中有一个或多个缺失。
| 基因 | 类型 | 结果 | 位置 在有丝分裂基因组中 |
结构 |
|---|---|---|---|---|
| MT-ATP8 | 蛋白质编码 | 三磷酸腺苷合酶,Fo亚基8(复合物ⅴ) | 08,366–08,572 (与MT-ATP6重叠) | L |
| MT-ATP6 | 蛋白质编码 | 三磷酸腺苷合酶,Fo亚基6(复合物ⅴ) | 08,527–09,207 (与MT-ATP8重叠) | L |
| MT-CO1 | 蛋白质编码 | 细胞色素c氧化酶亚单位1(复合物ⅳ) | 05,904–07,445 | L |
| MT-CO2 | 蛋白质编码 | 细胞色素c氧化酶,亚单位2(复合物ⅳ) | 07,586–08,269 | L |
| MT-CO3 | 蛋白质编码 | 细胞色素c氧化酶,亚单位3(复合物ⅳ) | 09,207–09,990 | L |
| MT-CYB | 蛋白质编码 | 细胞色素b(复合物三) | 14,747–15,887 | L |
| MT-ND1 | 蛋白质编码 | NADH脱氢酶,亚单位1(复合物1) | 03,307–04,262 | L |
| MT-ND2 | 蛋白质编码 | NADH脱氢酶,亚单位2(复合物1) | 04,470–05,511 | L |
| MT-ND3 | 蛋白质编码 | NADH脱氢酶,亚基3(复合物1) | 10,059–10,404 | L |
| MT-ND4L | 蛋白质编码 | NADH脱氢酶,亚单位4L(复合物ⅰ) | 10,470–10,766 (与MT-ND4重叠) | L |
| MT-ND4 | 蛋白质编码 | NADH脱氢酶,亚基4(复合物1) | 10,760–12,137 (与MT-ND4L重叠) | L |
| MT-ND5 | 蛋白质编码 | NADH脱氢酶,亚单位5(复合物1) | 12,337–14,148 | L |
| MT-ND6 | 蛋白质编码 | NADH脱氢酶,亚基6(复合物1) | 14,149–14,673 | H |
| MT-RNR2 | 蛋白质编码 | Humanin | — | — |
| MT-TA | 转移核糖核酸 | tRNA-丙氨酸(丙氨酸或丙氨酸) | 05,587–05,655 | H |
| MT-TR | 转移核糖核酸 | tRNA-精氨酸(精氨酸或精氨酸) | 10,405–10,469 | L |
| MT-TN | 转移核糖核酸 | tRNA-天冬酰胺 | 05,657–05,729 | H |
| MT-TD | 转移核糖核酸 | tRNA-天冬氨酸(Asp或D) | 07,518–07,585 | L |
| MT-TC | 转移核糖核酸 | tRNA-半胱氨酸 | 05,761–05,826 | H |
| MT-TE | 转移核糖核酸 | tRNA-谷氨酸(谷氨酸或谷氨酸) | 14,674–14,742 | H |
| MT-TQ | 转移核糖核酸 | 谷氨酰胺 | 04,329–04,400 | H |
| MT-TG | 转移核糖核酸 | tRNA-甘氨酸(甘氨酸或甘氨酸) | 09,991–10,058 | L |
| MT-TH | 转移核糖核酸 | tRNA-组氨酸 | 12,138–12,206 | L |
| MT-TI | 转移核糖核酸 | tRNA-异亮氨酸(异亮氨酸或异亮氨酸) | 04,263–04,331 | L |
| MT-TL1 | 转移核糖核酸 | tRNA-亮氨酸(亮氨酸或亮氨酸) | 03,230–03,304 | L |
| MT-TL2 | 转移核糖核酸 | tRNA-亮氨酸(亮CUN或亮) | 12,266–12,336 | L |
| MT-TK | 转移核糖核酸 | tRNA-赖氨酸(赖氨酸或赖氨酸) | 08,295–08,364 | L |
| MT-TM | 转移核糖核酸 | tRNA-甲硫氨酸(蛋氨酸或蛋氨酸) | 04,402–04,469 | L |
| MT-TF | 转移核糖核酸 | tRNA-苯丙氨酸(苯丙氨酸或苯丙氨酸) | 00,577–00,647 | L |
| MT-TP | 转移核糖核酸 | tRNA-脯氨酸(脯氨酸或脯氨酸) | 15,956–16,023 | H |
| MT-TS1 | 转移核糖核酸 | tRNA-丝氨酸(丝氨酸-UCN或丝氨酸) | 07,446–07,514 | H |
| MT-TS2 | 转移核糖核酸 | tRNA-丝氨酸(丝氨酸-AGY或丝氨酸) | 12,207–12,265 | L |
| MT-TT | 转移核糖核酸 | tRNA-苏氨酸(苏氨酸或苏氨酸) | 15,888–15,953 | L |
| MT-TW | 转移核糖核酸 | tRNA-色氨酸(色氨酸或钨) | 05,512–05,579 | L |
| MT-TY | 转移核糖核酸 | tRNA-酪氨酸(酪氨酸或酪氨酸) | 05,826–05,891 | H |
| MT-TV | 转移核糖核酸 | tRNA-缬氨酸(缬氨酸或缬氨酸) | 01,602–01,670 | L |
| MT-RNR1 | 核糖体核糖核酸 | 小亚单位:SSU (12S) | 00,648–01,601 | L |
| MT-RNR2 | 核糖体核糖核酸 | 大亚单位:LSU (16S) | 01,671–03,229 | L |
3.3 在植物中
真菌和植物中mtDNA基因的含量和大小存在很大差异,尽管似乎所有真核生物中都存在核心基因子集(除了少数完全没有线粒体的基因)[40]。一些植物物种有巨大的线粒体基因组,mtDNA二裂蝇子草含有多达11,300,000个碱基对[40]。令人惊讶的是,即使是那些巨大的mtDNA,也含有与具有小得多的mtdna的相关植物相同数量和种类的基因[41]。黄瓜线粒体的基因组由三条环状染色体组成(长度分别为1556、84和45千碱基),它们在复制方面完全或很大程度上是自主的[42]。
3.4 在原生生物中
4 基因组多样性编辑
线粒体基因组中有六种主要的基因组类型,根据它们的结构(例如圆形与线形)、大小、内含子或质粒样结构的存在以及遗传物质是单一分子还是同质或异质分子的集合进行分类[45]。
4.1 动物
动物细胞中只有一种线粒体基因组类型。这种基因组通常包含一个环形分子,其遗传物质(1型)的含量在11-28 kbp之间[45]。
4.2 植物和真菌
在植物和真菌中发现了三种不同的基因组类型。第一种类型是环状基因组,含有内含子(类型2),长度范围为19-1000 kbp。第二种基因组类型是环状基因组(约20-1000 kbp),也具有质粒样结构(1 kb)(类型3)。在植物和真菌中可以找到的最终基因组类型是由同源的脱氧核糖核酸分子组成的线性基因组(类型5)。
4.3 原生生物
原生生物包含最多样的线粒体基因组,在这个王国发现了五种不同的类型。植物和真菌基因组中提到的2型、3型和5型也存在于一些原生生物中,两种独特的基因组类型也是如此。这些独特类型中的一种是环状脱氧核糖核酸分子的异质集合(类型4),而另一种是线性分子的异质集合(类型6)。基因组类型4和6的大小各为1-200 kbp。
内共生基因转移,即线粒体基因组中编码的基因转移到细胞主基因组的过程,可能解释了为什么像人类这样更复杂的生物比像原生生物这样更简单的生物具有更小的线粒体基因组。
| 基因组类型[45] | 类型 | 内含子 | 大小 | 形状 | 描述 |
|---|---|---|---|---|---|
| 1 | 动物 | 不 | 11–28 kbp | 环形 | 单分子 |
| 2 | 真菌,植物,原生动物 | 是 | 19–1000 kbp | 环形 | 单分子 |
| 3 | 真菌,植物,原生动物 | 不 | 20–1000 kbp | 环形 | 大分子和小质粒样结构 |
| 4 | 原生生物 | 不 | 1–200 kbp | 环形 | 异质分子群 |
| 5 | 真菌,植物,原生动物 | 不 | 1–200 kbp | 线性的 | 同质分子群 |
| 6 | 原生生物 | 不 | 1–200 kbp | 线性的 | 异质分子群 |
5 复制编辑
线粒体脱氧核糖核酸由脱氧核糖核酸聚合酶γ复合物复制,该复合物由一种由聚乙二醇基因编码的140千道尔顿催化脱氧核糖核酸聚合酶和两种由聚乙二醇基因编码的55千道尔顿辅助亚基组成[46]。复制子机制由脱氧核糖核酸聚合酶、闪烁蛋白和线粒体单边带蛋白组成。TINKING是一个解旋酶,在5 '到3 '方向展开dsDNA的一小段[47]。所有这些多肽都编码在核基因组中。
胚胎发生期间,mtDNA的复制从受精卵母细胞到植入前胚胎受到严格的下调[48]。mtDNA每细胞拷贝数的减少在线粒体瓶颈中发挥了作用,利用细胞间的变异性来改善破坏性突变的遗传[49]。在胚泡阶段,mtDNA复制的开始是滋养外胚层细胞特有的[48]。相反,内部细胞团的细胞限制mtDNA复制,直到它们接收到分化为特定细胞类型的信号[48]。
6 转录编辑
7 突变和疾病编辑
7.1 敏感性
mtDNA因其邻近呼吸道而特别容易受到呼吸链产生的活性氧的影响,这一概念仍有争议[51]。 mtDNA积累的氧化碱基损伤不比核脱氧核糖核酸多[52]。据报道,至少某些类型的氧化脱氧核糖核酸损伤在线粒体中比在细胞核中修复得更有效[53]。 mtDNA包装的蛋白质似乎和核染色质的蛋白质一样具有保护作用[54]。此外,线粒体进化出一种独特的机制,通过降解过度受损的基因组,然后复制完整/修复的mtDNA,来维持mtDNA的完整性。这种机制不存在于细胞核中,并且由线粒体中存在的mtDNA的多个拷贝来实现[55]。mtDNA突变的结果可能是某些蛋白质编码指令的改变[56],这可能对机体代谢和/或健康有影响。
7.2 遗传疾病
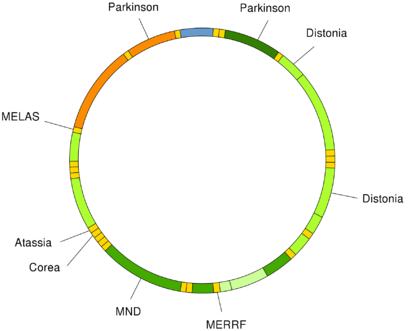
线粒体DNA突变可导致多种疾病,包括运动不耐症和卡恩斯-塞尔综合征(KSS),这会导致一个人丧失心脏、眼睛和肌肉运动的全部功能。一些证据表明,它们可能是衰老过程和与年龄相关的病理的主要原因[57]。特别是在疾病的情况下,细胞中突变mtDNA分子的比例被称为异源性。异源性的细胞内和细胞间分布决定了疾病的发作和严重程度 [58],并受到细胞内和发育过程中复杂随机过程的影响[59][59]。
线粒体tRNAs的突变可能导致像MELAS和MERRF综合征这样的严重疾病。
编码线粒体所用蛋白质的核基因突变也可能导致线粒体疾病。这些疾病不遵循线粒体遗传模式,而是遵循孟德尔遗传模式[60]。
7.3 用于疾病诊断
最近,mtDNA的一个突变被用来帮助前列腺活检阴性的患者诊断前列腺癌[61][62]。
7.4 与衰老的关系
尽管这个想法有争议,但一些证据表明衰老和线粒体基因组功能障碍之间有联系[63]。本质上,mtDNA的突变打破了活性氧产生和酶清除活性氧(通过超氧化物歧化酶、过氧化氢酶、谷胱甘肽过氧化物酶等酶)之间的平衡。然而,一些增加蠕虫体内活性氧产生的突变(例如,通过降低抗氧化防御)会增加而不是减少蠕虫的寿命[51]。此外,裸鼹鼠,老鼠大小的啮齿类动物,寿命是老鼠的八倍,尽管与老鼠相比,它们的抗氧化防御能力降低,对生物分子的氧化损伤增加[64]。曾经,人们认为有一个正反馈回路在起作用(“恶性循环”);随着线粒体脱氧核糖核酸积累自由基引起的遗传损伤,线粒体失去功能,自由基泄漏到细胞质中。线粒体功能下降会降低整体代谢效率[65]。 然而,这一概念最终被证明是不成立的,因为它证明了被基因改变以加速积累mtDNA突变的小鼠确实会过早衰老,但是它们的组织不会像“恶性循环”假说所预测的那样产生更多的活性氧[66]。支持寿命和线粒体基因之间的联系,一些研究发现线粒体基因的生化特性和物种的寿命之间有关联[67]。正在进行广泛的研究,以进一步调查这一联系和对抗衰老的方法。目前,基因治疗和营养补充是正在进行的研究的热门领域[68][69]。Bjelakovic等人分析了1977年至2012年间共涉及296,707名参与者的78项研究结果,并得出结论,抗氧化剂补充剂既不能降低全因死亡率,也不能延长寿命,而其中一些,如β胡萝卜素、维生素E和更高剂量的维生素A,实际上可能会增加死亡率[70]。
7.5 神经退行性疾病
mtDNA损伤增加是几种神经退行性疾病的特征。
阿尔茨海默氏症患者的大脑在核基因和mtDNA的氧化基因损伤水平都有所提高,但是mtDNA的水平比核基因高大约10倍[71]。有人提出衰老的线粒体是阿尔茨海默病中神经退行性变的关键因素[72]。
在亨廷顿氏病中,突变亨廷顿蛋白引起线粒体功能障碍,包括抑制线粒体电子转运、活性氧水平升高和氧化应激增加[73]。突变亨廷顿蛋白促进对mtDNA以及核脱氧核糖核酸的氧化损伤,这可能导致亨廷顿氏病病理[74]。
脱氧核糖核酸氧化产物8-氧代鸟嘌呤(8-氧代)是一个公认的氧化脱氧核糖核酸损伤的标记。肌萎缩侧索硬化患者通常修复脊髓运动神经元mtDNA区8-氧代脱氧核糖核酸损伤的酶受损[75]。 因此,运动神经元对mtDNA的氧化损伤可能是肌萎缩侧索硬化症病因中的一个重要因素。
7.6 mtDNA碱基组成与动物寿命的相关性
在过去的十年里,由瓦迪姆·弗赖菲尔德教授领导的以色列研究小组已经表明,mtDNA碱基的组成和特定动物物种的最大寿命之间存在着非常强而显著的相关性[76][77][78]。正如他们的工作所证明的那样,mtDNA鸟嘌呤+胞嘧啶含量越高,动物物种的最长寿命就越长。另一个惊人的发现是,mtDNA(CG%)与最大寿命的相关性独立于众所周知的动物物种代谢率与最大寿命之间的相关性。mtDNA气相色谱%和静息代谢率以倍增的方式解释了动物物种最大寿命的差异(即物种最大寿命=它们的mtDNA(CG%) *代谢率)[77]。为了支持科学界对mtDNA特征和动物寿命进行比较分析,建立了一个名为MitoAge的专门数据库[79]。
7.7 与非典型脱氧核糖核酸结构的关系
缺失断点经常出现在显示非典型(非B)构象的区域内或附近,即发夹、十字形和苜蓿叶形元素[80]。此外,有数据支持扭曲螺旋的内在弯曲区域和长G-4分体参与引发不稳定事件。此外,在GC偏斜区域和简并序列基序YMMYMNNMMHM的附近一致观察到较高的断点密度[81]。最近(2017年)发现,迄今为止测序的所有有丝分裂基因组都含有十字形脱氧核糖核酸形成所需的许多反向重复序列,这些基因座在复制起点、D环和茎环中特别丰富[82]。
8 用于识别编辑
核基因是从父母双方遗传的,基因在重组过程中会重新排列,与此不同,mtDNA的母体和后代之间通常没有变化。尽管mtDNA也重组,但它是通过在同一线粒体内复制自己来重组的。正因为如此,也因为动物mtDNA的突变率高于核基因的突变率[83], mtDNA是通过女性(母系血统)追踪祖先的有力工具,并被用于追踪数百代以来许多物种的祖先。
(动物的)快速突变率使mtDNA有助于评估一个物种内个体或群体的遗传关系,也有助于识别和量化不同物种之间的系统发育。为此,生物学家确定并比较来自不同个体或物种的mtDNA序列。来自比较的数据被用于构建序列之间的关系网络,该网络提供了对从中提取mtDNAs的个体或物种之间的关系的估计。mtDNA可以用来估计近亲和远亲物种之间的关系。由于mtDNA在动物中的高突变率,密码子的第三个位置变化相对较快,从而提供了关于密切相关的个体或物种之间遗传距离的信息。另一方面,mt蛋白的取代率非常低,因此氨基酸变化积累缓慢(在第一和第二密码子位置有相应的缓慢变化),因此它们提供了关于远亲物种遗传距离的信息。因此,分别处理密码子位置间置换率的统计模型可用于同时估计包含近缘和远缘物种的系统发育[84]。
1996年,在田纳西州诉保罗·韦尔一案中,线粒体DNA首次被美国法庭接受为证据[84]。
在1998年美国法院审理的宾夕法尼亚联邦诉[帕特里夏·林恩·罗勒案中[85],线粒体DNA第一次被宾夕法尼亚州接受为证据[86][87]。该案件出现在真实犯罪剧系列《法医档案》(第五季)第五季第55集。
线粒体脱氧核糖核酸首次被美国加州接纳为证据,是在2002年大卫·韦斯特菲尔德在圣地亚哥成功起诉7岁的丹妮尔·范·达姆(Danielle van Dam)绑架和谋杀案中:线粒体脱氧核糖核酸被用于人类和狗的身份识别[88]。这是美国第一次接受犬类基因的试验[89]。
理查三世国王的遗体是通过比较他的mtDNA和他姐姐的两个母系后裔来鉴定的[90]。
9 历史编辑
10 线粒体序列数据库编辑
已经建立了几个专门的数据库来收集线粒体基因组序列和其他信息。尽管它们中的大部分集中在序列数据上,但也有一些包含系统发育或功能信息。
11 线粒体突变数据库编辑
有几个专门的数据库报告了人类线粒体基因的多态性和突变,以及对其致病性的评估。
- 缓解措施:对导致人类线粒体蛋白编码基因非同义替换的所有核苷酸变化的预先计算的致病性预测的集合 [3]。
- 有丝分裂图:人类线粒体基因多态性和突变的概要[4]。
参考文献
- [1]
^Sykes B (10 September 2003). "Mitochondrial DNA and human history". The Human Genome. Wellcome Trust. Archived from the original on 7 September 2015. Retrieved 5 February 2012..
- [2]
^"Mitochondrial DNA: The Eve Gene". Bradshaw Foundation. Bradshaw Foundation. Retrieved 5 November 2012..
- [3]
^Avise, John C. (2000). Phylogeography: The history and formation of species. Harvard University Press. ISBN 978-0674666382..
- [4]
^Luo S, Valencia CA, Zhang J, Lee NC, Slone J, Gui B, Wang X, Li Z, Dell S, Brown J, Chen SM, Chien YH, Hwu WL, Fan PC, Wong LJ, Atwal PS, Huang T (December 2018). "Biparental Inheritance of Mitochondrial DNA in Humans". Proceedings of the National Academy of Sciences of the United States of America. 115 (51): 13039–13044. doi:10.1073/pnas.1810946115. PMC 6304937. PMID 30478036..
- [5]
^Boursot P, Bonhomme F (1986-01-01). "[Not Available]". Génétique, Sélection, Évolution. 18 (1): 73–98. doi:10.1186/1297-9686-18-1-73. PMC 2713894. PMID 22879234..
- [6]
^Delsuc F, Stanhope MJ, Douzery EJ (August 2003). "Molecular systematics of armadillos (Xenarthra, Dasypodidae): contribution of maximum likelihood and Bayesian analyses of mitochondrial and nuclear genes". Molecular Phylogenetics and Evolution. 28 (2): 261–75. doi:10.1016/s1055-7903(03)00111-8. PMID 12878463..
- [7]
^Hassanin A, An J, Ropiquet A, Nguyen TT, Couloux A (March 2013). "Combining multiple autosomal introns for studying shallow phylogeny and taxonomy of Laurasiatherian mammals: Application to the tribe Bovini (Cetartiodactyla, Bovidae)". Molecular Phylogenetics and Evolution. 66 (3): 766–75. doi:10.1016/j.ympev.2012.11.003. PMID 23159894..
- [8]
^Wiesner RJ, Rüegg JC, Morano I (March 1992). "Counting target molecules by exponential polymerase chain reaction: copy number of mitochondrial DNA in rat tissues". Biochemical and Biophysical Research Communications. 183 (2): 553–9. doi:10.1016/0006-291X(92)90517-O. PMID 1550563..
- [9]
^van der Giezen M, Tovar J, Clark CG (2005). "Mitochondrion‐Derived Organelles in Protists and Fungi". A Survey of Cell Biology. International Review of Cytology. 244. pp. 175–225. doi:10.1016/S0074-7696(05)44005-X. ISBN 978-0-12-364648-4. PMID 16157181..
- [10]
^Adams KL, Palmer JD (December 2003). "Evolution of mitochondrial gene content: gene loss and transfer to the nucleus". Molecular Phylogenetics and Evolution. 29 (3): 380–95. doi:10.1016/S1055-7903(03)00194-5. PMID 14615181..
- [11]
^Björkholm P, Harish A, Hagström E, Ernst AM, Andersson SG (August 2015). "Mitochondrial genomes are retained by selective constraints on protein targeting". Proceedings of the National Academy of Sciences of the United States of America. 112 (33): 10154–61. Bibcode:2015PNAS..11210154B. doi:10.1073/pnas.1421372112. PMC 4547212. PMID 26195779..
- [12]
^Allen JF (August 2015). "Why chloroplasts and mitochondria retain their own genomes and genetic systems: Colocation for redox regulation of gene expression". Proceedings of the National Academy of Sciences of the United States of America. 112 (33): 10231–8. Bibcode:2015PNAS..11210231A. doi:10.1073/pnas.1500012112. PMC 4547249. PMID 26286985..
- [13]
^Wolff JN, Gemmell NJ (November 2008). "Lost in the zygote: the dilution of paternal mtDNA upon fertilization". Heredity. 101 (5): 429–34. doi:10.1038/hdy.2008.74. PMID 18685570..
- [14]
^Gabriel MS, Chan SW, Alhathal N, Chen JZ, Zini A (August 2012). "Influence of microsurgical varicocelectomy on human sperm mitochondrial DNA copy number: a pilot study". Journal of Assisted Reproduction and Genetics. 29 (8): 759–64. doi:10.1007/s10815-012-9785-z. PMC 3430774. PMID 22562241..
- [15]
^Sutovsky P, Moreno RD, Ramalho-Santos J, Dominko T, Simerly C, Schatten G (November 1999). "Ubiquitin tag for sperm mitochondria". Nature. 402 (6760): 371–2. Bibcode:1999Natur.402..371S. doi:10.1038/46466. PMID 10586873. Discussed in: Travis, John (2000). "Mom's Eggs Execute Dad's Mitochondria". Science News. 157 (1): 5. doi:10.2307/4012086. JSTOR 4012086. Archived from the original on 19 December 2007..
- [16]
^"Hiring a DNA Testing Company Genealogy". Family Search. The Church of Jesus Christ of Latter-day Saints. 2016. Retrieved 2016-10-02..
- [17]
^Vilà C, Savolainen P, Maldonado JE, Amorim IR, Rice JE, Honeycutt RL, Crandall KA, Lundeberg J, Wayne RK (June 1997). "Multiple and ancient origins of the domestic dog". Science. 276 (5319): 1687–9. doi:10.1126/science.276.5319.1687. PMID 9180076..
- [18]
^Wolff JN, White DJ, Woodhams M, White HE, Gemmell NJ (2011). "The strength and timing of the mitochondrial bottleneck in salmon suggests a conserved mechanism in vertebrates". PLOS ONE. 6 (5): e20522. Bibcode:2011PLoSO...620522W. doi:10.1371/journal.pone.0020522. PMC 3105079. PMID 21655224..
- [19]
^Cree LM, Samuels DC, de Sousa Lopes SC, Rajasimha HK, Wonnapinij P, Mann JR, Dahl HH, Chinnery PF (February 2008). "A reduction of mitochondrial DNA molecules during embryogenesis explains the rapid segregation of genotypes". Nature Genetics. 40 (2): 249–54. doi:10.1038/ng.2007.63. PMID 18223651..
- [20]
^Cao L, Shitara H, Horii T, Nagao Y, Imai H, Abe K, Hara T, Hayashi J, Yonekawa H (March 2007). "The mitochondrial bottleneck occurs without reduction of mtDNA content in female mouse germ cells". Nature Genetics. 39 (3): 386–90. doi:10.1038/ng1970. PMID 17293866..
- [21]
^Wai T, Teoli D, Shoubridge EA (December 2008). "The mitochondrial DNA genetic bottleneck results from replication of a subpopulation of genomes". Nature Genetics. 40 (12): 1484–8. doi:10.1038/ng.258. PMID 19029901..
- [22]
^Alexander M, Ho SY, Molak M, Barnett R, Carlborg Ö, Dorshorst B, Honaker C, Besnier F, Wahlberg P, Dobney K, Siegel P, Andersson L, Larson G (October 2015). "Mitogenomic analysis of a 50-generation chicken pedigree reveals a rapid rate of mitochondrial evolution and evidence for paternal mtDNA inheritance". Biology Letters. 11 (10): 20150561. doi:10.1098/rsbl.2015.0561. PMC 4650172. PMID 26510672..
- [23]
^Gyllensten U, Wharton D, Josefsson A, Wilson AC (July 1991). "Paternal inheritance of mitochondrial DNA in mice". Nature. 352 (6332): 255–7. Bibcode:1991Natur.352..255G. doi:10.1038/352255a0. PMID 1857422..
- [24]
^Shitara H, Hayashi JI, Takahama S, Kaneda H, Yonekawa H (February 1998). "Maternal inheritance of mouse mtDNA in interspecific hybrids: segregation of the leaked paternal mtDNA followed by the prevention of subsequent paternal leakage". Genetics. 148 (2): 851–7. PMC 1459812. PMID 9504930..
- [25]
^Zhao X, Li N, Guo W, Hu X, Liu Z, Gong G, Wang A, Feng J, Wu C (October 2004). "Further evidence for paternal inheritance of mitochondrial DNA in the sheep (Ovis aries)". Heredity. 93 (4): 399–403. doi:10.1038/sj.hdy.6800516. PMID 15266295..
- [26]
^Steinborn R, Zakhartchenko V, Jelyazkov J, Klein D, Wolf E, Müller M, Brem G (April 1998). "Composition of parental mitochondrial DNA in cloned bovine embryos". FEBS Letters. 426 (3): 352–6. doi:10.1016/S0014-5793(98)00350-0. PMID 9600265..
- [27]
^Schwartz M, Vissing J (August 2002). "Paternal inheritance of mitochondrial DNA". The New England Journal of Medicine. 347 (8): 576–80. doi:10.1056/NEJMoa020350. PMID 12192017..
- [28]
^Luo S, Valencia CA, Zhang J, Lee NC, Slone J, Gui B, Wang X, Li Z, Dell S, Brown J, Chen SM, Chien YH, Hwu WL, Fan PC, Wong LJ, Atwal PS, Huang T (December 2018). "Biparental Inheritance of Mitochondrial DNA in Humans". Proceedings of the National Academy of Sciences of the United States of America. 115 (51): 13039–13044. doi:10.1073/pnas.1810946115. PMID 30478036..
- [29]
^Passamonti M, Ghiselli F (February 2009). "Doubly uniparental inheritance: two mitochondrial genomes, one precious model for organelle DNA inheritance and evolution". DNA and Cell Biology. 28 (2): 79–89. doi:10.1089/dna.2008.0807. PMID 19196051..
- [30]
^Kondo R, Matsuura ET, Chigusa SI (April 1992). "Further observation of paternal transmission of Drosophila mitochondrial DNA by PCR selective amplification method". Genetical Research. 59 (2): 81–4. doi:10.1017/S0016672300030287. PMID 1628820..
- [31]
^Wolff JN, Nafisinia M, Sutovsky P, Ballard JW (January 2013). "Paternal transmission of mitochondrial DNA as an integral part of mitochondrial inheritance in metapopulations of Drosophila simulans". Heredity. 110 (1): 57–62. doi:10.1038/hdy.2012.60. PMC 3522233. PMID 23010820..
- [32]
^Meusel MS, Moritz RF (December 1993). "Transfer of paternal mitochondrial DNA during fertilization of honeybee (Apis mellifera L.) eggs". Current Genetics. 24 (6): 539–43. doi:10.1007/BF00351719. PMID 8299176..
- [33]
^Fontaine KM, Cooley JR, Simon C (September 2007). "Evidence for paternal leakage in hybrid periodical cicadas (Hemiptera: Magicicada spp.)". PLOS ONE. 2 (9): e892. Bibcode:2007PLoSO...2..892F. doi:10.1371/journal.pone.0000892. PMC 1963320. PMID 17849021..
- [34]
^Frith M (14 October 2003). "Ban on scientists trying to create three-parent baby". The Independent..
- [35]
^Roberts M (2016-09-27). "First 'three person baby' born using new method". BBC News (in 英语). Retrieved 2016-09-28..
- [36]
^Nosek J, Tomáska L, Fukuhara H, Suyama Y, Kovác L (May 1998). "Linear mitochondrial genomes: 30 years down the line". Trends in Genetics. 14 (5): 184–8. doi:10.1016/S0168-9525(98)01443-7. PMID 9613202..
- [37]
^Balaresque P, Bowden GR, Adams SM, Leung HY, King TE, Rosser ZH, Goodwin J, Moisan JP, Richard C, Millward A, Demaine AG, Barbujani G, Previderè C, Wilson IJ, Tyler-Smith C, Jobling MA (January 2010). "A predominantly neolithic origin for European paternal lineages". PLoS Biology. 8 (1): e1000285. doi:10.1371/journal.pbio.1000285. PMC 2799514. PMID 20087410..
- [38]
^Barroso Lima NC, Prosdocimi F (March 2018). "The heavy strand dilemma of vertebrate mitochondria on genome sequencing age: number of encoded genes or G + T content?". Mitochondrial DNA. Part A, DNA Mapping, Sequencing, and Analysis. 29 (2): 300–302. doi:10.1080/24701394.2016.1275603. PMID 28129726..
- [39]
^Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–65. Bibcode:1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534..
- [40]
^Johnston IG, Williams BP (February 2016). "Evolutionary Inference across Eukaryotes Identifies Specific Pressures Favoring Mitochondrial Gene Retention". Cell Systems. 2 (2): 101–11. doi:10.1016/j.cels.2016.01.013. PMID 27135164..
- [41]
^Ward BL, Anderson RS, Bendich AJ (September 1981). "The mitochondrial genome is large and variable in a family of plants (cucurbitaceae)". Cell. 25 (3): 793–803. doi:10.1016/0092-8674(81)90187-2. PMID 6269758..
- [42]
^Alverson AJ, Rice DW, Dickinson S, Barry K, Palmer JD (July 2011). "Origins and recombination of the bacterial-sized multichromosomal mitochondrial genome of cucumber". The Plant Cell. 23 (7): 2499–513. doi:10.1105/tpc.111.087189. JSTOR 41433488. PMC 3226218. PMID 21742987..
- [43]
^"Mitochondrial DNA (mtDNA)" (PDF). Integrated DNA Technologies..
- [44]
^Tyagi S, Pande V, Das A (February 2014). "Whole mitochondrial genome sequence of an Indian Plasmodium falciparum field isolate". The Korean Journal of Parasitology (in English). 52 (1): 99–103. doi:10.3347/kjp.2014.52.1.99. PMC 3949004. PMID 24623891.CS1 maint: Unrecognized language (link).
- [45]
^Kolesnikov AA, Gerasimov ES (December 2012). "Diversity of mitochondrial genome organization". Biochemistry. Biokhimiia. 77 (13): 1424–35. doi:10.1134/S0006297912130020. PMID 23379519..
- [46]
^Yakubovskaya E, Chen Z, Carrodeguas JA, Kisker C, Bogenhagen DF (January 2006). "Functional human mitochondrial DNA polymerase gamma forms a heterotrimer". The Journal of Biological Chemistry. 281 (1): 374–82. doi:10.1074/jbc.M509730200. PMID 16263719..
- [47]
^Jemt E, Farge G, Bäckström S, Holmlund T, Gustafsson CM, Falkenberg M (November 2011). "The mitochondrial DNA helicase TWINKLE can assemble on a closed circular template and support initiation of DNA synthesis". Nucleic Acids Research. 39 (21): 9238–49. doi:10.1093/nar/gkr653. PMC 3241658. PMID 21840902..
- [48]
^St John JC, Facucho-Oliveira J, Jiang Y, Kelly R, Salah R (2010). "Mitochondrial DNA transmission, replication and inheritance: a journey from the gamete through the embryo and into offspring and embryonic stem cells". Human Reproduction Update. 16 (5): 488–509. doi:10.1093/humupd/dmq002. PMID 20231166..
- [49]
^Falkenberg M, Larsson NG, Gustafsson CM (2007-06-19). "DNA replication and transcription in mammalian mitochondria". Annual Review of Biochemistry. 76 (1): 679–99. doi:10.1146/annurev.biochem.76.060305.152028. PMID 17408359..
- [50]
^Ojala D, Montoya J, Attardi G (April 1981). "tRNA punctuation model of RNA processing in human mitochondria". Nature. 290 (5806): 470–4. Bibcode:1981Natur.290..470O. doi:10.1038/290470a0. PMID 7219536..
- [51]
^Alexeyev MF (October 2009). "Is there more to aging than mitochondrial DNA and reactive oxygen species?". The FEBS Journal. 276 (20): 5768–87. doi:10.1111/j.1742-4658.2009.07269.x. PMC 3097520. PMID 19796285..
- [52]
^Anson RM, Hudson E, Bohr VA (February 2000). "Mitochondrial endogenous oxidative damage has been overestimated". FASEB Journal. 14 (2): 355–60. doi:10.1096/fasebj.14.2.355. PMID 10657991..
- [53]
^Thorslund T, Sunesen M, Bohr VA, Stevnsner T (April 2002). "Repair of 8-oxoG is slower in endogenous nuclear genes than in mitochondrial DNA and is without strand bias". DNA Repair. 1 (4): 261–73. doi:10.1016/S1568-7864(02)00003-4. PMID 12509245..
- [54]
^Guliaeva NA, Kuznetsova EA, Gaziev AI (2006). "[Proteins associated with mitochondrial DNA protect it against the action of X-rays and hydrogen peroxide]" [Proteins associated with mitochondrial DNA protect it against the action of X-rays and hydrogen peroxide]. Biofizika (in Russian). 51 (4): 692–7. PMID 16909848.CS1 maint: Unrecognized language (link).
- [55]
^Alexeyev M, Shokolenko I, Wilson G, LeDoux S (May 2013). "The maintenance of mitochondrial DNA integrity--critical analysis and update". Cold Spring Harbor Perspectives in Biology. 5 (5): a012641. doi:10.1101/cshperspect.a012641. PMC 3632056. PMID 23637283..
- [56]
^Hogan CM (2010). "Mutation". In Monosson E, Cleveland CJ. Encyclopedia of Earth. Washington DC: National Council for Science and the Environment..
- [57]
^Alexeyev MF, Ledoux SP, Wilson GL (October 2004). "Mitochondrial DNA and aging". Clinical Science. 107 (4): 355–64. doi:10.1042/CS20040148. PMID 15279618..
- [58]
^Burgstaller JP, Johnston IG, Poulton J (January 2015). "Mitochondrial DNA disease and developmental implications for reproductive strategies". Molecular Human Reproduction. 21 (1): 11–22. doi:10.1093/molehr/gau090. PMC 4275042. PMID 25425607..
- [59]
^Johnston IG, Burgstaller JP, Havlicek V, Kolbe T, Rülicke T, Brem G, Poulton J, Jones NS (June 2015). "Stochastic modelling, Bayesian inference, and new in vivo measurements elucidate the debated mtDNA bottleneck mechanism". eLife. 4: e07464. doi:10.7554/eLife.07464. PMC 4486817. PMID 26035426..
- [60]
^Angelini C, Bello L, Spinazzi M, Ferrati C (July 2009). "Mitochondrial disorders of the nuclear genome". Acta Myologica. 28 (1): 16–23. PMC 2859630. PMID 19772191..
- [61]
^Reguly B, Jakupciak JP, Parr RL (October 2010). "3.4 kb mitochondrial genome deletion serves as a surrogate predictive biomarker for prostate cancer in histopathologically benign biopsy cores". Canadian Urological Association Journal. 4 (5): E118–22. PMC 2950771. PMID 20944788..
- [62]
^Robinson K, Creed J, Reguly B, Powell C, Wittock R, Klein D, Maggrah A, Klotz L, Parr RL, Dakubo GD (June 2010). "Accurate prediction of repeat prostate biopsy outcomes by a mitochondrial DNA deletion assay". Prostate Cancer and Prostatic Diseases. 13 (2): 126–31. doi:10.1038/pcan.2009.64. PMID 20084081..
- [63]
^de Grey A (1999). The Mitochondrial Free Radical Theory of Aging (PDF). ISBN 978-1-57059-564-6.[页码请求].
- [64]
^Lewis KN, Andziak B, Yang T, Buffenstein R (October 2013). "The naked mole-rat response to oxidative stress: just deal with it". Antioxidants & Redox Signaling. 19 (12): 1388–99. doi:10.1089/ars.2012.4911. PMC 3791056. PMID 23025341..
- [65]
^Shigenaga MK, Hagen TM, Ames BN (November 1994). "Oxidative damage and mitochondrial decay in aging". Proceedings of the National Academy of Sciences of the United States of America. 91 (23): 10771–8. Bibcode:1994PNAS...9110771S. doi:10.1073/pnas.91.23.10771. JSTOR 2365473. PMC 45108. PMID 7971961..
- [66]
^Trifunovic A, Hansson A, Wredenberg A, Rovio AT, Dufour E, Khvorostov I, Spelbrink JN, Wibom R, Jacobs HT, Larsson NG (December 2005). "Somatic mtDNA mutations cause aging phenotypes without affecting reactive oxygen species production". Proceedings of the National Academy of Sciences of the United States of America. 102 (50): 17993–8. Bibcode:2005PNAS..10217993T. doi:10.1073/pnas.0508886102. JSTOR 4152716. PMC 1312403. PMID 16332961..
- [67]
^Aledo JC, Li Y, de Magalhães JP, Ruíz-Camacho M, Pérez-Claros JA (April 2011). "Mitochondrially encoded methionine is inversely related to longevity in mammals". Aging Cell. 10 (2): 198–207. doi:10.1111/j.1474-9726.2010.00657.x. PMID 21108730..
- [68]
^Ferrari CK (2004). "Functional foods, herbs and nutraceuticals: towards biochemical mechanisms of healthy aging". Biogerontology. 5 (5): 275–89. doi:10.1007/s10522-004-2566-z. PMID 15547316..
- [69]
^Taylor RW (February 2005). "Gene therapy for the treatment of mitochondrial DNA disorders". Expert Opinion on Biological Therapy. 5 (2): 183–94. doi:10.1517/14712598.5.2.183. PMID 15757380..
- [70]
^Bjelakovic G, Nikolova D, Gluud C (September 2013). "Antioxidant supplements to prevent mortality". JAMA. 310 (11): 1178–9. doi:10.1001/jama.2013.277028. PMID 24045742..
- [71]
^Wang J, Xiong S, Xie C, Markesbery WR, Lovell MA (May 2005). "Increased oxidative damage in nuclear and mitochondrial DNA in Alzheimer's disease". Journal of Neurochemistry. 93 (4): 953–62. doi:10.1111/j.1471-4159.2005.03053.x. PMID 15857398..
- [72]
^Bonda DJ, Wang X, Lee HG, Smith MA, Perry G, Zhu X (April 2014). "Neuronal failure in Alzheimer's disease: a view through the oxidative stress looking-glass". Neuroscience Bulletin. 30 (2): 243–52. doi:10.1007/s12264-013-1424-x. PMC 4097013. PMID 24733654..
- [73]
^Liu Z, Zhou T, Ziegler AC, Dimitrion P, Zuo L (2017). "Oxidative Stress in Neurodegenerative Diseases: From Molecular Mechanisms to Clinical Applications". Oxidative Medicine and Cellular Longevity. 2017: 2525967. doi:10.1155/2017/2525967. PMC 5529664. PMID 28785371..
- [74]
^Ayala-Peña S (September 2013). "Role of oxidative DNA damage in mitochondrial dysfunction and Huntington's disease pathogenesis". Free Radical Biology & Medicine. 62: 102–110. doi:10.1016/j.freeradbiomed.2013.04.017. PMC 3722255. PMID 23602907..
- [75]
^Kikuchi H, Furuta A, Nishioka K, Suzuki SO, Nakabeppu Y, Iwaki T (April 2002). "Impairment of mitochondrial DNA repair enzymes against accumulation of 8-oxo-guanine in the spinal motor neurons of amyotrophic lateral sclerosis". Acta Neuropathologica. 103 (4): 408–14. doi:10.1007/s00401-001-0480-x. PMID 11904761..
- [76]
^Lehmann G, Budovsky A, Muradian KK, Fraifeld VE (2006). "Mitochondrial genome anatomy and species-specific lifespan". Rejuvenation Research. 9 (2): 223–6. doi:10.1089/rej.2006.9.223. PMID 16706648..
- [77]
^Lehmann G, Segal E, Muradian KK, Fraifeld VE (April 2008). "Do mitochondrial DNA and metabolic rate complement each other in determination of the mammalian maximum longevity?". Rejuvenation Research. 11 (2): 409–17. doi:10.1089/rej.2008.0676. PMID 18442324..
- [78]
^Lehmann G, Muradian KK, Fraifeld VE (2013). "Telomere length and body temperature-independent determinants of mammalian longevity?". Frontiers in Genetics. 4 (111): 111. doi:10.3389/fgene.2013.00111. PMC 3680702. PMID 23781235..
- [79]
^Toren D, Barzilay T, Tacutu R, Lehmann G, Muradian KK, Fraifeld VE (January 2016). "MitoAge: a database for comparative analysis of mitochondrial DNA, with a special focus on animal longevity". Nucleic Acids Research. 44 (D1): D1262–5. doi:10.1093/nar/gkv1187. PMC 4702847. PMID 26590258..
- [80]
^Damas J, Carneiro J, Gonçalves J, Stewart JB, Samuels DC, Amorim A, Pereira F (September 2012). "Mitochondrial DNA deletions are associated with non-B DNA conformations". Nucleic Acids Research. 40 (16): 7606–21. doi:10.1093/nar/gks500. PMC 3439893. PMID 22661583..
- [81]
^Oliveira PH, da Silva CL, Cabral JM (2013). "An appraisal of human mitochondrial DNA instability: new insights into the role of non-canonical DNA structures and sequence motifs". PLOS ONE. 8 (3): e59907. Bibcode:2013PLoSO...859907O. doi:10.1371/journal.pone.0059907. PMC 3612095. PMID 23555828..
- [82]
^Cechová J, Lýsek J, Bartas M, Brázda V (April 2018). "Complex analyses of inverted repeats in mitochondrial genomes revealed their importance and variability". Bioinformatics. 34 (7): 1081–1085. doi:10.1093/bioinformatics/btx729. PMC 6030915. PMID 29126205..
- [83]
^Brown WM, George M, Wilson AC (April 1979). "Rapid evolution of animal mitochondrial DNA". Proceedings of the National Academy of Sciences of the United States of America. 76 (4): 1967–71. Bibcode:1979PNAS...76.1967B. doi:10.1073/pnas.76.4.1967. JSTOR 69636. PMC 383514. PMID 109836..
- [84]
^Taylor RW, Turnbull DM (May 2005). "Mitochondrial DNA mutations in human disease". Nature Reviews. Genetics. 6 (5): 389–402. doi:10.1038/nrg1606. PMC 1762815. PMID 15861210..
- [85]
^[1] Court case name listed in the appeal.[full citation needed] Retrieved 17 April 2015..
- [86]
^[2][self-published source] Defense lawyer. Retrieved 17 April 2015..
- [87]
^Garlicki, Debbie (11 March 1998). "DNA Tests Got Rorrer Life In Jail". The Morning Call..
- [88]
^"Judge allows DNA in Samantha Runnion case," Associated Press, 18 February 2005. Retrieved 4 April 2007..
- [89]
^"Canine DNA Admitted In California Murder Case," Pit Bulletin Legal News, 5 December 2013. Retrieved 21 January 2014..
- [90]
^Kennedy, Maev (4 February 2013). "Richard III: DNA confirms twisted bones belong to king". The Guardian. Retrieved 7 December 2014..
- [91]
^Nass MM, Nass S (December 1963). "INTRAMITOCHONDRIAL FIBERS WITH DNA CHARACTERISTICS. I. FIXATION AND ELECTRON STAINING REACTIONS". The Journal of Cell Biology. 19 (3): 593–611. doi:10.1083/jcb.19.3.593. PMC 2106331. PMID 14086138..
- [92]
^Schatz G, Haslbrunner E, Tuppy H (March 1964). "DEOXYRIBONUCLEIC ACID ASSOCIATED WITH YEAST MITOCHONDRIA". Biochemical and Biophysical Research Communications. 15 (2): 127–32. doi:10.1016/0006-291X(64)90311-0. PMID 26410904..
- [93]
^Ehler E, Novotný J, Juras A, Chylenski M, Moravcík O, Paces J (January 2019). "AmtDB: a database of ancient human mitochondrial genomes". Nucleic Acids Research. 47 (D1): D29–D32. doi:10.1093/nar/gky843. PMC 6324066. PMID 30247677..
- [94]
^Gu Z, Li J, Gao S, Gong M, Wang J, Xu H, Zhang C, Wang J (June 2011). "InterMitoBase: an annotated database and analysis platform of protein-protein interactions for human mitochondria". BMC Genomics. 12: 335. doi:10.1186/1471-2164-12-335. PMC 3142533. PMID 21718467..
- [95]
^Damas J, Carneiro J, Amorim A, Pereira F (January 2014). "MitoBreak: the mitochondrial DNA breakpoints database". Nucleic Acids Research. 42 (Database issue): D1261–8. doi:10.1093/nar/gkt982. PMC 3965124. PMID 24170808..
- [96]
^Iwasaki W, Fukunaga T, Isagozawa R, Yamada K, Maeda Y, Satoh TP, Sado T, Mabuchi K, Takeshima H, Miya M, Nishida M (November 2013). "MitoFish and MitoAnnotator: a mitochondrial genome database of fish with an accurate and automatic annotation pipeline". Molecular Biology and Evolution. 30 (11): 2531–40. doi:10.1093/molbev/mst141. PMC 3808866. PMID 23955518..
- [97]
^Cawthorn DM, Steinman HA, Witthuhn RC (November 2011). "Establishment of a mitochondrial DNA sequence database for the identification of fish species commercially available in South Africa". Molecular Ecology Resources. 11 (6): 979–91. doi:10.1111/j.1755-0998.2011.03039.x. PMID 21689383..
- [98]
^Lee YS, Oh J, Kim YU, Kim N, Yang S, Hwang UW (January 2008). "Mitome: dynamic and interactive database for comparative mitochondrial genomics in metazoan animals". Nucleic Acids Research. 36 (Database issue): D938–42. doi:10.1093/nar/gkm763. PMC 2238945. PMID 17940090..
- [99]
^Catalano D, Licciulli F, Turi A, Grillo G, Saccone C, D'Elia D (January 2006). "MitoRes: a resource of nuclear-encoded mitochondrial genes and their products in Metazoa". BMC Bioinformatics. 7: 36. doi:10.1186/1471-2105-7-36. PMC 1395343. PMID 16433928..
- [100]
^Kumar M, Kapil A, Shanker A (November 2014). "MitoSatPlant: mitochondrial microsatellites database of viridiplantae". Mitochondrion. 19 Pt B: 334–7. doi:10.1016/j.mito.2014.02.002. PMID 24561221..
- [101]
^D'Onorio de Meo P, D'Antonio M, Griggio F, Lupi R, Borsani M, Pavesi G, Castrignanò T, Pesole G, Gissi C (January 2012). "MitoZoa 2.0: a database resource and search tools for comparative and evolutionary analyses of mitochondrial genomes in Metazoa". Nucleic Acids Research. 40 (Database issue): D1168–72. doi:10.1093/nar/gkr1144. PMC 3245153. PMID 22123747..
暂无